
喜讯!易基因表观转录组学RNA-BS技术服务见刊《核酸研究》
2024年2月15日,吉林大学张涛、赵飞宇、李金泽为共同第一作者,吉林大学李占军、隋婷婷及赖良学为共同通讯在《Nucleic Acids Research》(NAR/ IF14.9)发表题为“Programmable RNA 5-methylcytosine (m5C) modification of cellular RNAs by dCasRx conjugated methyltransferase and demethylase”的研究论文,通过m5C RNA-BS等分析揭示RCMS(reengineered m5C modification system)编辑系统能够精确地在特定RNA位点掺入或去除m5C修饰,改变细胞转录本的稳定性。此外,RCMS编辑系统还调控tRNA m5C水平,影响细胞的转录本丰度、细胞增殖和迁移能力。易基因科技为本研究提供mRNA BS-seq建库测序分析技术服务。

标题:Programmable RNA 5-methylcytosine (m5C) modification of cellular RNAs by dCasRx conjugated methyltransferase and demethylase(通过dCasRx偶联的甲基转移酶和去甲基化酶实现细胞RNA的可编程m5C修饰)
时间:2024.2.15
期刊:Nucleic Acids Research(核酸研究)
影响因子:IF 14.9

研究摘要:
5-甲基胞嘧啶(m5C)是一种丰富的RNA修饰,在调控RNA命运和基因表达中起着至关重要的作用。尽管最近在理解m5C的生物学作用方面取得了进展,但无法在转录本中的特异性位点引入m5C阻碍了揭示特异性m5C与表型结果之间直接联系的研究。本研究作者开发了一种基于CRISPR-Cas13d的编辑系统,名为重编程m5C修饰系统(reengineered m5C modification system,简称RCMS),用于特定转录本中的靶向m5C甲基化和去甲基化。RCMS编辑系统由核定位的dCasRx与甲基转移酶(NSUN2/NSUN6)或去甲基化酶(小鼠Tet2的催化结构域)(ten-eleven translocation 2,TET-2)结合而成,可以在精确m5C位点操纵甲基化事件。研究揭示了RCMS编辑系统可以诱导特异性m5C甲基化和去甲基化。此外研究还证实了RCMS编辑系统在调控转运RNA m5C水平以及通过m5C介导的机制诱导转录本丰度和细胞增殖变化的能力。研究结果共同将RCMS编辑系统确立为一种靶向表观转录组编辑工具,有助于鉴定单基因m5C变化及其可能产生结果。对于深入理解RNA修饰在生物学过程中的作用以及开发新的治疗策略具有重要意义。
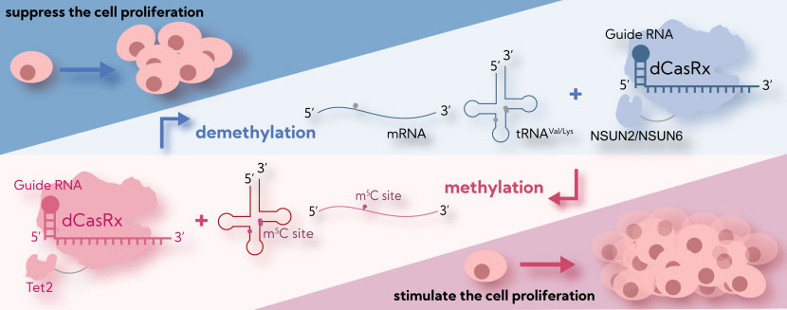
图形摘要
研究结果:
(1)可编程m5C编辑系统设计
l NSUN6酶验证可编程位点特异性m5C writer活性
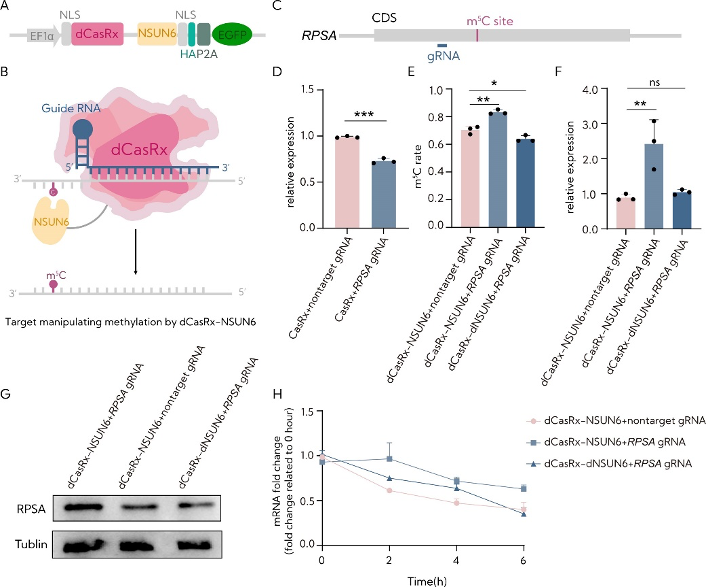
图1:NSUN6在HEK293T细胞中验证可编程位点特异性m5C writer活性。
A. 将NSUN6甲基转移酶与dCasRx结合以构建RCMS-dCasRx-NSUN6编辑器。
B. RCMS策略。该策略涉及使用与NSUN6甲基转移酶结合的可编程RNA结合蛋白,如dCasRx,通过靶转录本中特定gRNA方式,诱导从C到m5C的位点特异性甲基化。
C. RPSA mRNA中m5C位点和gRNA靶向区域示意图。
D. 通过RT-qPCR使用CasRx评估RPSA gRNA的靶向效率(n=3)。
E. 使用HiTOM深度序列分析评估RCMS dCasRx-NSUN6编辑器靶向m5C甲基化后RPSA m5C位点的m5C比率水平(n=3)。
F. 在HEK293T细胞中,使用与非靶向gRNA或RPSA gRNA结合的dCasRx-NSUN6或dCasRx-dNSUN6评估RPSA mRNA水平(n=3)。
G. 用dCasRx-NSUN6或dCasRx-dNSUN6联合非靶向gRNA或RPSA gRNA分析HEK293T细胞中RPSA的蛋白水平。
H. 在用10 mg/ml放线菌素D(一种有效的转录抑制剂)处理的HEK293T细胞中,通过RT-qPCR检查指定时间点的RPSA水平(n = 3)。
“非靶向gRNA”被用作非靶向对照。误差线表示平均值±SD。*P<0.05,**P<0.01,***P<0.001,n.s.表示双尾不成对双样本t检验不显著。
l dCasRx-NSUN2在人类细胞中对内源性转录本的甲基化
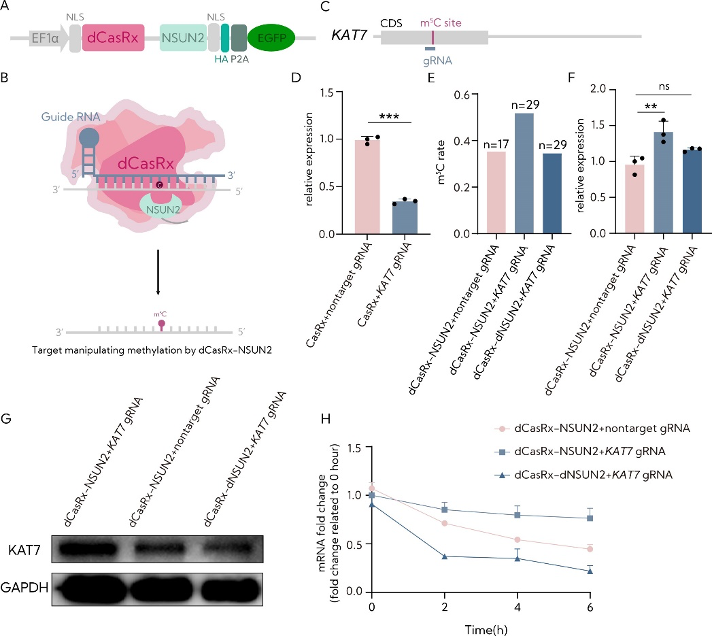
图2:dCasRx-NSUN2在人类细胞中对内源性转录本的甲基化。
A. 将NSUN2甲基转移酶分别与dCasRx结合以生成RCMS dCasRx-NSUN2编辑器。
B. 提出带有NSUN2的dCasRx RCMSs策略。
C. 展示KAT7 mRNA中m5C位点和gRNA靶向区域示意图。
D. RT-qPCR检测KAT7 gRNA使用CasRx的靶向效率(n=3)。
E. 通过RCMS dCasRx-NSUN2编辑器对靶向m5C甲基化后KAT7 m5C位点的C-to-T突变率进行定量,每个条形图上方显示总测试数[dCasRx-Tet2 CD+非靶向gRNA(6/17),dCasRx-Tet2 CD+KAT7 gRNA(15/29),dCasRx-Tet2 CD+KAT7 gRNA(10/29);n1/n2,n1表示胞嘧啶数量,n2表示总测试数。
F. HEK293T细胞中,使用与非靶向gRNA或KAT7 gRNA结合的dCasRx-NSUN2分析KAT7 mRNA水平(n=3)。
G. 用dCasRx-NSUN2或dCasRx-dNSUN2联合非靶向gRNA或KAT7 gRNA评估HEK293T细胞中KAT7的蛋白水平。
H. 在用10 mg/ml放线菌素D(一种强效的转录抑制剂)处理的HEK293T细胞中,通过RT-qPCR分析指定时间点的KAT7 mRNA水平(n = 3)。
“非靶向gRNA”用于非靶向。误差线表示平均值±SD。*P<0.05,**P<0.01,***P<0.001,n.s.表示双尾不成对双样本t检验(n=3)不显著。
l 通过Tet2验证可编程位点特异性m5C eraser活力
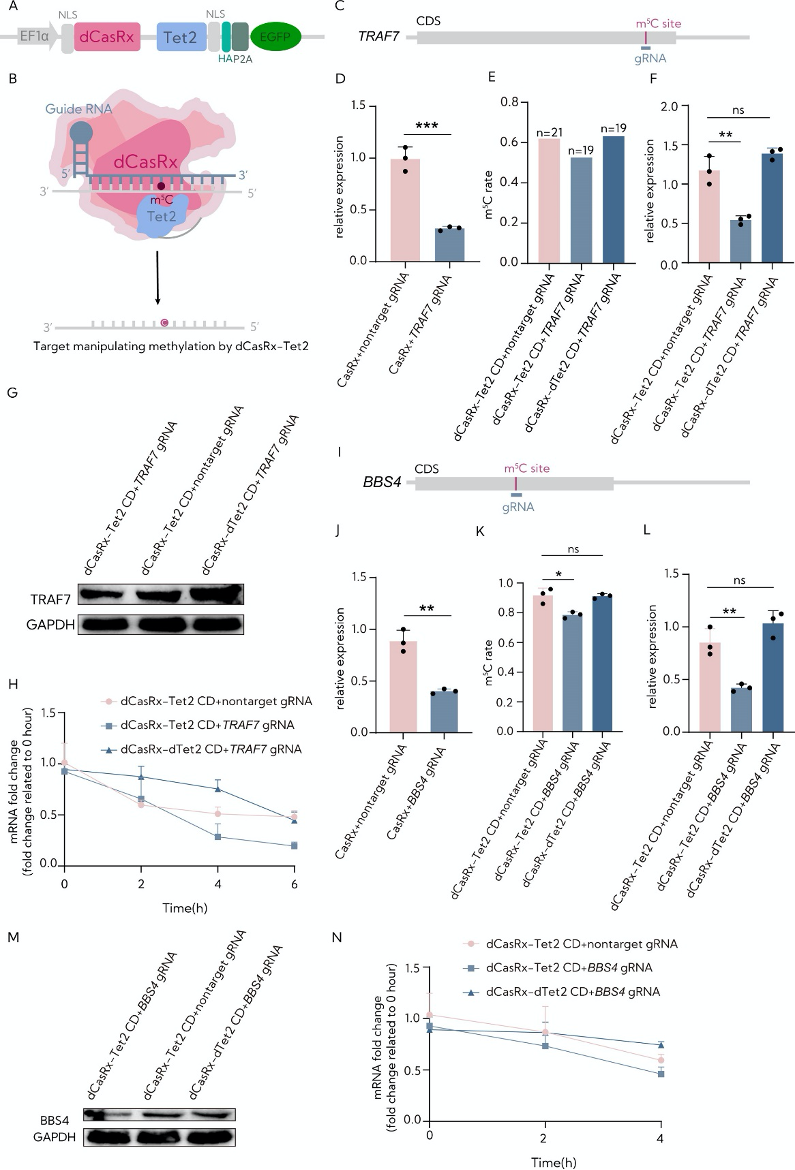
图3:通过RCMS eraser dCasRx-Tet2 CD进行mRNA转录本的靶向去甲基化。
A. 将Tet2 CD(Tet2催化域)分别与dCasRx结合以生成RCMS dCasRx-Tet2 CD编辑器。
B. 提出带有Tet2 CD的dCasRx RCMSs策略。
C. TRAF7(肿瘤坏死因子TNF受体相关因子7)mRNA m5C位点和gRNA靶向区域示意图。
D. 通过RT-qPCR检测TRAF7 gRNA的CasRx靶向效率(n=3)。
E. 通过RCMS dCasRx-Tet2 CD编辑器靶向m5C甲基化后,对TRAF7 m5C位点的C-T突变比率进行定量检测,测试数字显示在每个条形图上方。dCasRx-Tet2 CD+非靶向gRNA(13/21),dCasRx-Tet2 CD+TRAF7 gRNA(10/19),dCasRx-Tet2 CD+TRAF7 gRNA(12/19);n1/n2,n1表示胞嘧啶数量,n2表示总检测数。
F. 使用与TRAF7 gRNA或非靶向gRNA结合的dCasRx-Tet2 CD分析TRAF7 mRNA水平(n = 3)。
G. 用dCasRx-Tet2 CD联合TRAF7 gRNA或非靶向gRNA检测HEK293T细胞中TRAF7的蛋白水平。
H. 在用10 mg/ml放线菌素D处理的HEK293T细胞中,通过RT-qPCR检测指定时间点的TRAF7 mRNA水平(n = 3)。
I. BBS4 (Bardet-Biedl综合征4)mRNA中m5C位点和gRNA靶向区域示意图。
J. 使用RT-qPCR检测BBS4 gRNA使用CasRx的靶向效率(n = 3)。
K. 使用HiTOM分析深度序列对dCasRx-Tet2CD编辑器靶向m5C甲基化后BBS4 m5C位点的m5C比率定量检测(n = 3)
L. 在HEK293T细胞中,使用与BBS4 gRNA或非靶向gRNA结合的dCasRx-Tet2CD分析BBS4 mRNA水平(n = 3)。
M. 用dCasRx-Tet2CD联合BBS4 gRNA或非靶向gRNA评估HEK293T细胞中BBS4的蛋白水平。
N. RT-qPCR检测10 mg/ml放线菌素D处理的HEK293T细胞在指定时间点的BBS4 mRNA水平(n=3)。
(2)RCMS在tRNA中诱导可编程位点特异性m5C水平
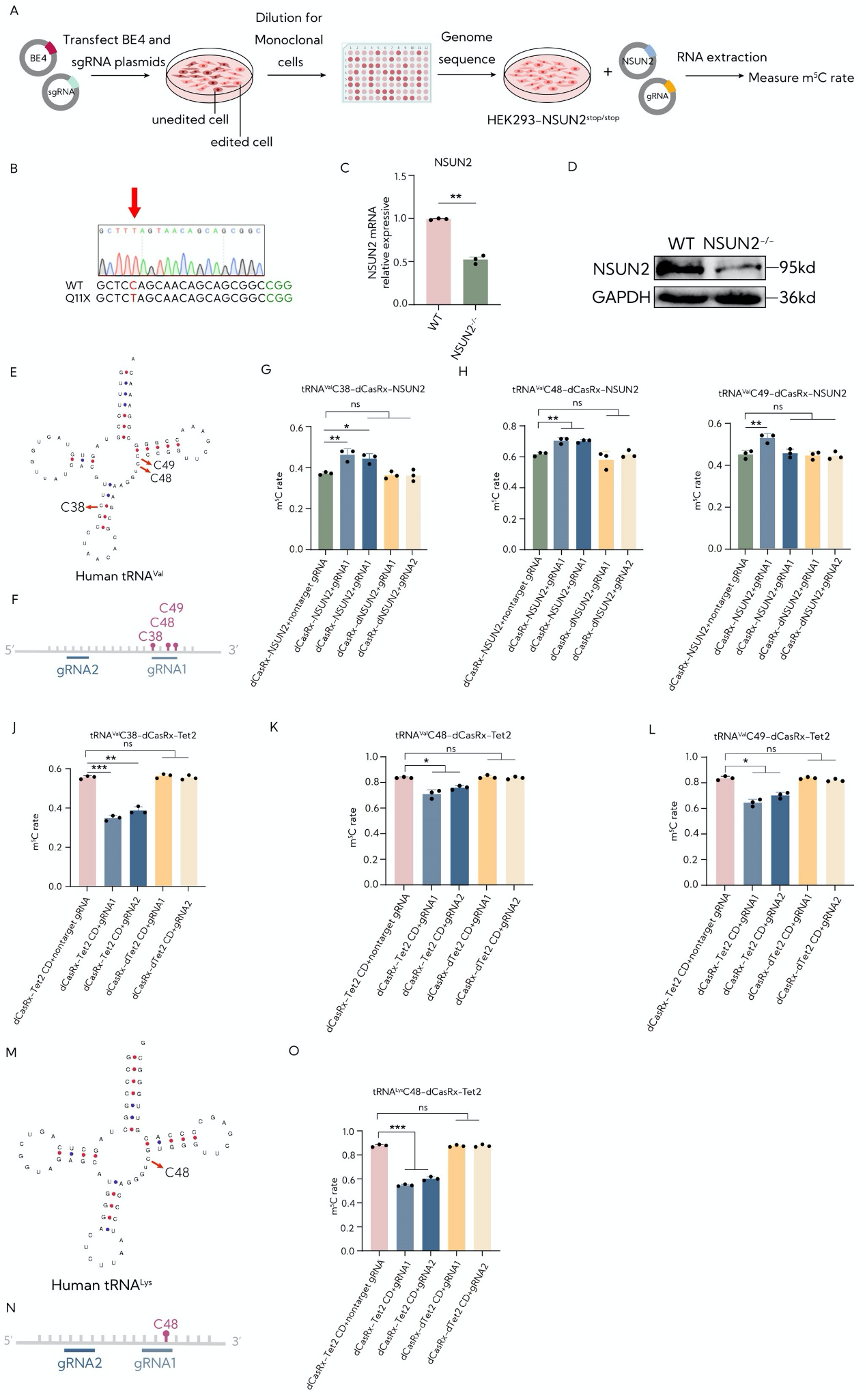
图4:HEK293T细胞中靶向处理tRNA转录本甲基化和去甲基化。
A. NSUN2-/-细胞系构建示意图。首先用编码BE4max编辑器和靶向NSUN2基因的gRNA的质粒转染HEK293T细胞。转染后,将细胞稀释并接种到96孔板中进行克隆选择,并筛选单克隆细胞的所需NSUN2-/-基因型。
B. Gln11中NSUN2-/-细胞系的Sanger测序色谱图。
C. NSUN2-/-细胞系中NSUN2 mRNA水平(n = 3)。
D. WT细胞和NSUN2-/-细胞中NSUN2蛋白表达的Western blot结果。
E. 人tRNAVal示意图,tRNAVal中三个m5C位点用红色箭头标记。
F. tRNAVal中gRNA位点示意图。
G-I. 使用dCasRx-NSUN2编辑器在NSUN2-/-细胞系中靶向m5C甲基化后,利用深度序列HiTOM分析tRNAVal的m5C38、m5C48和m5C49位点的m5C比率(n=3)。
J-L. 使用dCasRx-Tet2 CD编辑器在HEK293T细胞中靶向m5C甲基化后,利用深度序列HiTOM分析tRNAVal的m5C38、m5C48和m5C49位点的m5C比率(n=3)。
M. 人类tRNA Lys的示意图,tRNA Lys中的m5C48位点用红色箭头标记。
N. tRNA Lys中gRNA位置示意图。
O. 使用dCasRx-Tet2 CD编辑器在HEK293T细胞中靶向m5C甲基化后,利用深度序列HiTOM分析tRNA Lys的m5C48位点m5C比率(n=3)。
(3)RCMS编辑器在人类细胞中的非靶向甲基化(Off-target methylation)
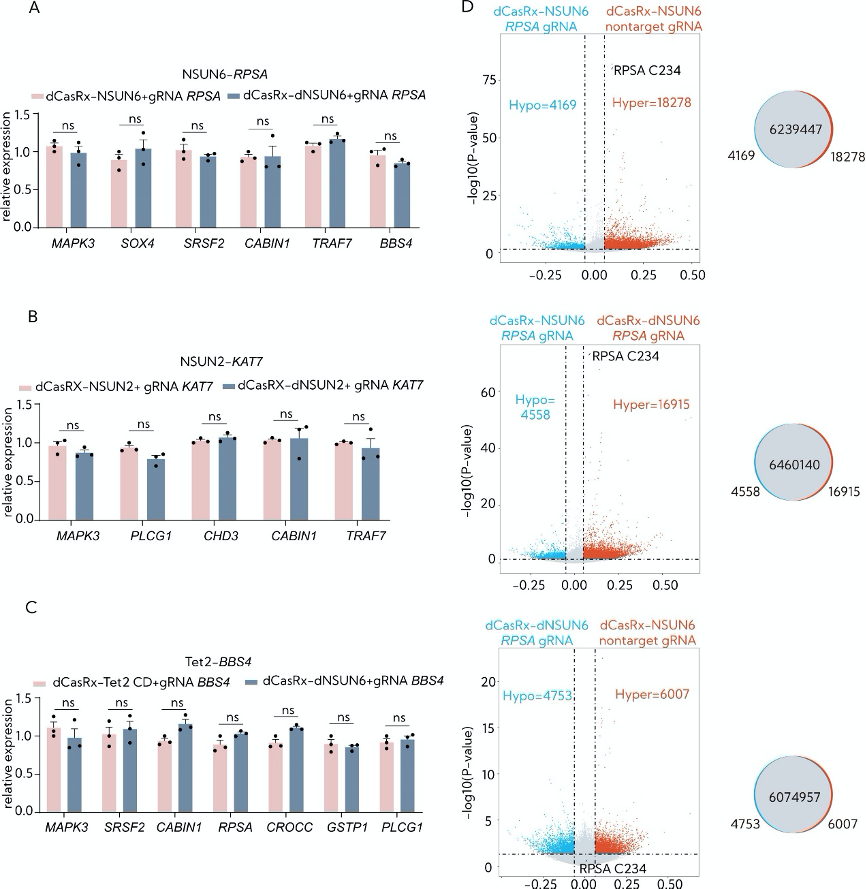
图5:RCMS dCasRx-NSUN6编辑器的特异性和非靶向甲基化。
A. 通过RCMS dCasRx-NSUN6编辑器靶向RPSA的m5C甲基化后,m5C富集转录本的mRNA水平(n = 3)。
B. 通过RCMS dCasRx-NSUN2编辑器靶向KAT7的m5C去甲基化后,m5C富集转录本的mRNA水平(n = 3)。
C. 用RCMS dCasRx-Tet2 CD编辑器靶向BBS4的m5C去甲基化后,m5C富集转录本的mRNA水平(n = 3)。
D. 用dCasRx-NSUN6或dCasRx-NSUN6和RPSA靶向gRNA或非靶向gRNA转导的HEK293T细胞中m5C甲基化位点的差异富集。顶部代表上述条件下m5C位点的差异甲基化,表明差异甲基化位点具有统计学意义(P<0.05)。底部代表维恩图,描绘了用于上述比较的所有甲基化m5C位点的重叠。用三个独立的生物学重复进行BS-seq分析。非靶向gRNA用于非靶向。误差线表示平均值±SD。*P<0.05,**P<0.01,***P<0.001,n.s.表示双向方差分析和Dunnett多重比较不显著。
(4)HepG2细胞中使用RCMS进行可编程RNA m5C修饰
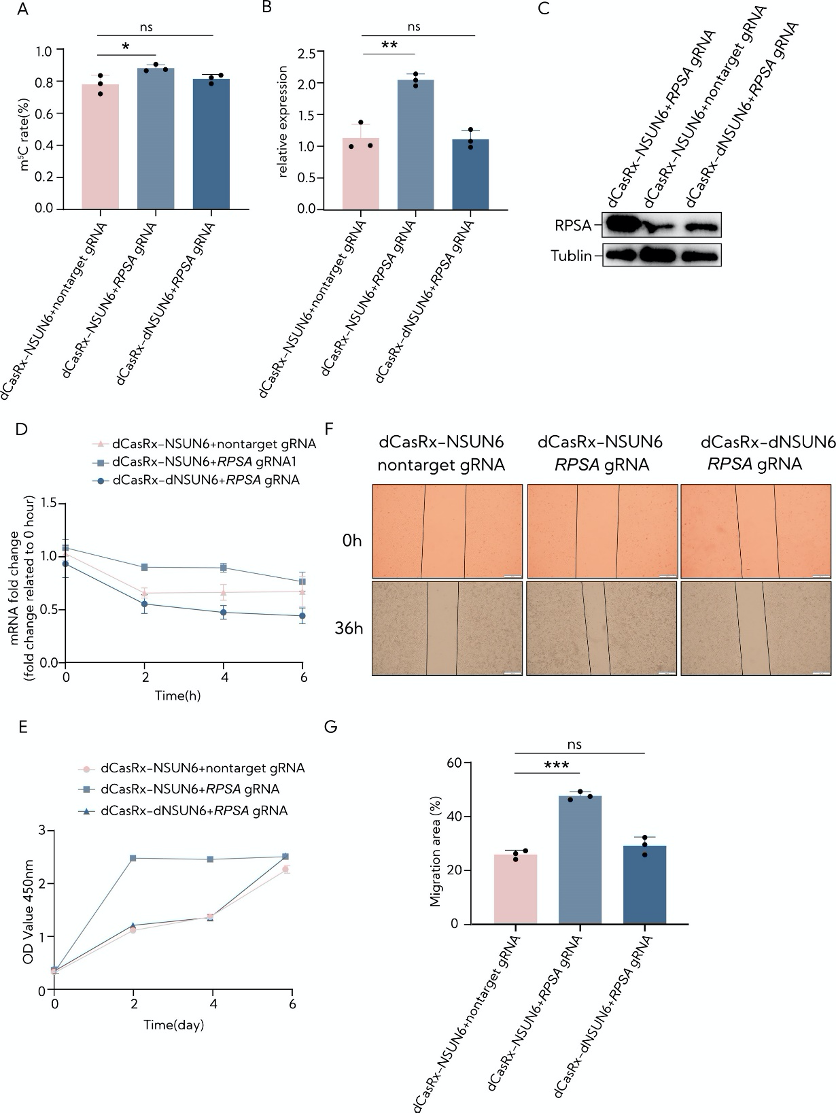
图6:RCMS dCasRx-NSUN6编辑器对HepG2细胞中的RPSA m5C位点进行修饰。
A. 深度序列HiTOM分析在RCMS dCasRx-NSUN6编辑器和RPSA gRNA-150 nt靶向下RPSA m5C234位点的m5C比率(n=3)。
B. dCasRx-NSUN6或dCasRx-dNSUN6联合非靶向gRNA或RPSA gRNA-150 nt转导的HEK293T细胞RPSA的mRNA水平(n = 3)。
C. 用dCasRx-NSUN6或dCasRx-dNSUN6联合非靶向gRNA或RPSA gRNA-150 nt转导的HEK293T细胞中RPSA的蛋白质水平。
D. 通过RT-qPCR在指定的时间点检测经10 mg/ml放线菌素D处理的HEK293T细胞中RPSA水平(n=3)。
E. CCK8法检测dCasRx-NSUN6或dCasRx-dNSUN6和非靶向gRNA或RPSA gRNA-150 nt转导的HepG2细胞增殖(n=6) 。
F. 使用dCasRx-NSUN6和RPSA gRNA-150 nt对RPSA进行靶向甲基化的HepG2细胞的划痕实验的代表性图像。在初始划痕后0h和36h记录划痕测量值。比例尺:200微米。
G. 伤口愈合试验的统计分析(n=3)。
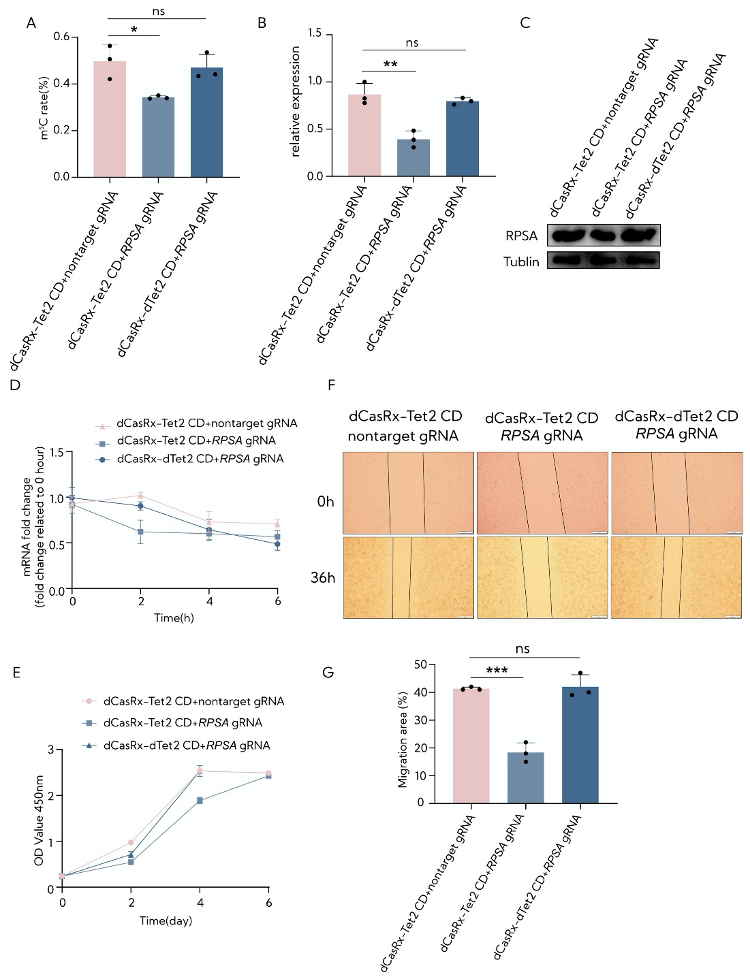
图7:RCMS dCasRx-NSUN6编辑器对HepG2细胞中的RPSA m5C位点进行修饰。
A. 利用深度序列HiTOM分析RCMS dCasRx-Tet2 CD编辑器和RPSA gRNA 0 nt靶向下RPSA m5C234位点的m5C比率(n=3) 。
B. dCasRx-Tet2 CD或dCas-dTet2 CD联合非靶向gRNA或RPSA gRNA 0 nt转导的HEK293T细胞RPSA的mRNA水平(n = 3)。
C. dCasRx-Tet2 CD或dCasRx-dTet2 CD联合非靶向gRNA或RPSA gRNA 0 nt转导的HepG2细胞RPSA的蛋白水平。
D. RT-qPCR在指定时间点检测经10 mg/ml放线菌素D处理的HepG2细胞中RPSA水平(n=3)。
E. CCK8法检测转染dCasRx-Tet2 CD或dCasRx-dTet2 CD和非靶向gRNA或RPSA gRNA 0 nt的HepG2细胞增殖(n=6)。
F. dCasRx-Tet2 CD和RPSA gRNA 0 nt对RPSA进行靶向甲基化的HepG2细胞中伤口愈合实验的代表性图像。在初始划痕后0和36小时记录划痕测量。比例尺:200微米。
G. 伤口愈合实验的统计分析。
研究小结
本研究标志着RCMS作为一种靶向表观转录组编辑工具的开创性建立,并探讨了其潜在应用。研究揭示了RCMS的广泛功能,允许在跨RNA靶标中进行精确的位点特异性m5C掺入或去除。这以甲基化依赖性方式改变了细胞转录本稳定性。研究新开发的RCMS具有特异性潜能可以阐明m5C与表型之间的功能关系,并推进m5C生物学的基础研究。
参考文献:
Zhang T, Zhao F, Li J, Sun X, Zhang X, Wang H, Fan P, Lai L, Li Z, Sui T. Programmable RNA 5-methylcytosine (m5C) modification of cellular RNAs by dCasRx conjugated methyltransferase and demethylase. Nucleic Acids Res. 2024 Feb 15. pii: 7608824. doi: 10.1093/nar/gkae110. PubMed PMID: 38366553.