
大家好,这里是专注表观组学十余年,领跑多组学科研服务的易基因。
近日,中山大学(深圳校区)农业与生物技术学院刘赛博博士等为第一作者,中山大学农业与生物技术学院黄晓辰副教授、中国海洋大学jian zhao为共同通讯作者,在纳米科学领域王牌期刊《ACS Nano》发表题为《Genome-Wide Molecular Adaptation in Algal Primary Productivity Induced by Prolonged Exposure to Environmentally Realistic Concentration of Nanoplastics》研究论文。研究探讨了纳米塑料(Nanoplastics, NPs)在环境相关浓度下对藻类(algae)初级生产力(Primary Productivity)的长期作用影响。研究人员选择三种不同电荷的聚苯乙烯纳米塑料(nPS、nPS-NH₂和nPS-COOH),以10 μg/L的环境相关浓度暴露于小球藻(Chlorella sp.)100天,通过表观基因组学(WGBS)和转录组学(RNA-seq)整合分析揭示了藻类对纳米塑料的表观遗传适应机制,为环境应激下的生物适应性研究提供新的方法和理论基础。深圳易基因提供的WGBS+RNA-seq技术助力阐明藻类对纳米塑料的适应性机制,包括DNA甲基化变化和基因表达调控。

标题:Genome-Wide Molecular Adaptation in Algal Primary Productivity Induced by Prolonged Exposure to Environmentally Realistic Concentration of Nanoplastics(由环境相关浓度的纳米塑料长期暴露引起的藻类初级生产力的全基因组分子适应)
时间:2024年10月19日
期刊:ACS Nano
影响因子:IF16/Q1
作者单位:中山大学深圳校区刘赛博、黄晓辰等
DOI:10.1021/acsnano.4c09709
本研究结果表明,藻类通过增加藻的数量(10.34–16.52%)、叶绿素a(Chl a)(11.28–17.65%)和碳固定酶活性(49.19–68.33%)适应了这三种类型的纳米塑料(nPS、nPS-NH₂和nPS-COOH),这一结果也通过自然水体培养实验得到了进一步证实。在个体水平上,基于藻类叶绿体数量和生物体积的分析,只有nPS导致藻类分化为两个异质亚群(54.92% vs 45.08%),而nPS-NH₂和nPS-COOH并未引起藻类群体分化。通过整合表观基因组学(WGBS)和转录组学(RNA-seq)揭示了藻类对纳米塑料的分子适应机制。藻类在暴露于nPS、nPS-NH₂和nPS-COOH时的平均甲基化率显著升高。与nPS和nPS-NH₂相比,暴露于nPS-COOH时通过差异甲基化区域(differentially methylated regions,DMRs)调控基因表达方向差异。本研究结果强调了评估纳米塑料长期生态毒性的重要性,并为理解纳米塑料对水生生态系统的影响提供有用信息。
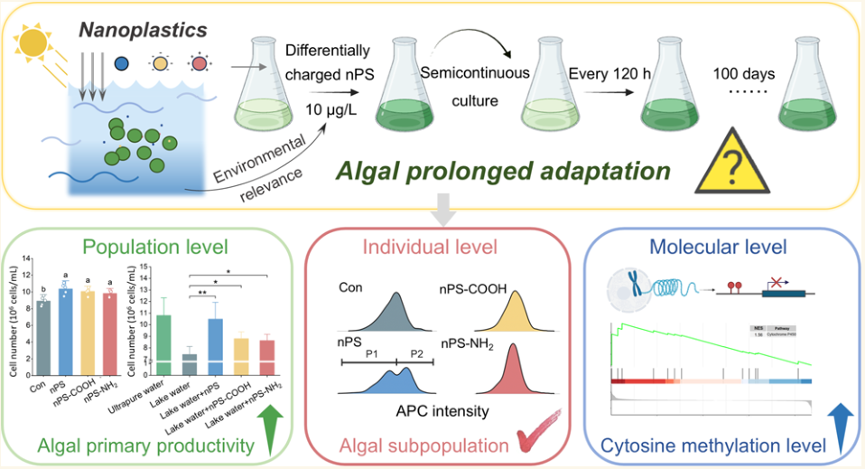
图形摘要
结果图形
(1)长期NPs暴露提升藻类初级生产力
在长达100天的纳米塑料(NPs)暴露实验中,研究人员发现藻类的初级生产力得到显著提升。藻类细胞数量在nPS、nPS-COOH和nPS-NH₂暴露组分别增加了16.50%、13.03%和10.32%。叶绿素a(Chl a)含量增加了11.28%-17.65%。然而,藻类生物量仅nPS和nPS-COOH暴露组增加,nPS-NH₂暴露组无显著变化。藻类的最大相对电子传递速率(rETRmax)在nPS和nPS-COOH暴露组中有所增加,且nPS-COOH暴露组的增加幅度最大。藻类通过光合作用固定二氧化碳的能力也得到了增强,这体现在所有三种类型的NPs均提高了RuBisCo活性(49.19%–68.33%)。研究人员还在自然水体中进行了暴露实验,结果表明,在经过100天的暴露后,nPS和nPS-COOH暴露组的藻类细胞数量、生物量和Chl a均有所增加,这与在超纯水制备的培养基中的实验结果一致。然而,nPS-NH₂暴露组的Chl a增加并不显著,这可能是由于nPS-NH₂与自然水体中的有机物结合,减少了与藻类细胞的接
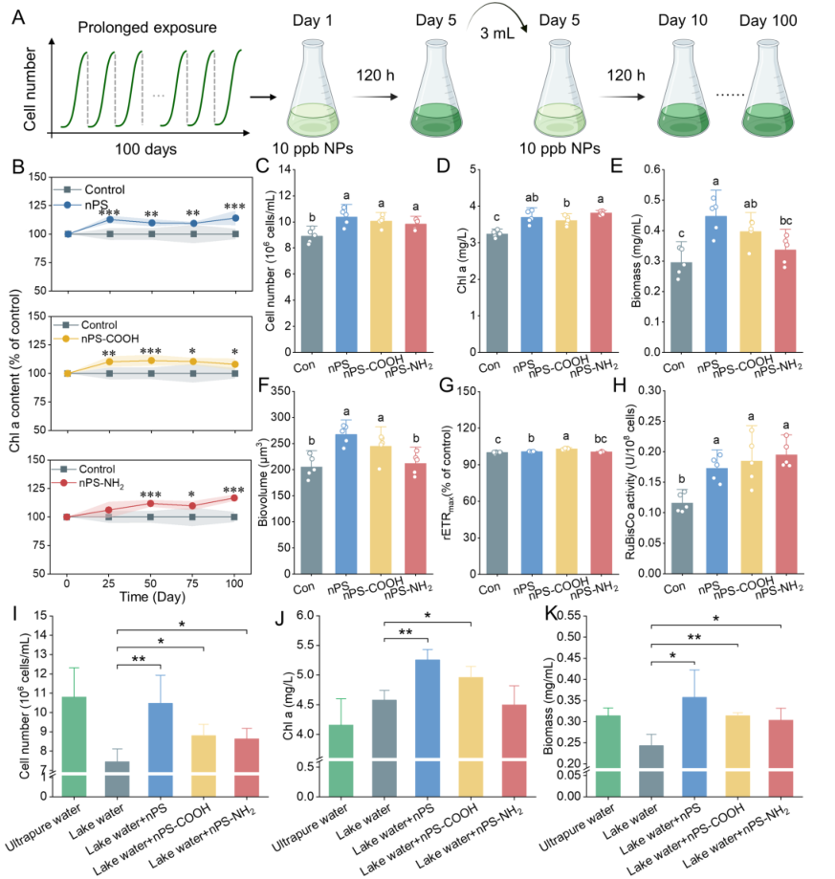
图1:藻类在长期适应纳米塑料(NPs)后的初级生产力。
(A) 藻类在实验室中适应NPs的实验设计图。
(B-G)藻类菌株在实验室适应期间的(B)叶绿素a(Chl a)含量、(C)藻类细胞数量、(D)叶绿素a含量、(E)生物量、(F)生物体积、(G)最大相对电子传递速率(rETRmax)。
(H-J)藻类在适应三种NPs 100天后的(H)RuBisCo活性、(I)藻类细胞数量、(J)叶绿素a含量。(K) 藻类在自然水体培养系统中暴露于三种NPs(100天)后的生物量。
(2)藻类初级生产力的个体性状分化
为了进一步揭示不同表面电荷的NPs对藻类初级生产力响应的差异,研究人员在个体水平上评估了藻类的叶绿体数量。结果显示,藻类细胞的平均叶绿体数量并未受到长期NPs适应的影响。尽管在三种NPs暴露组之间叶绿体体积没有显著差异,但在nPS暴露组中,从均质藻类群体中出现了两个具有低水平(P1)和高水平(P2)叶绿体表达的亚群。相比之下,nPS-COOH和nPS-NH₂暴露组并未表现出显著的分化现象。P1亚群(占总细胞数的54.92%)的平均信号强度显著下降至对照组的67.65%,而P2亚群(占总细胞数的45.08%)则显著增加至对照组的131.64%。研究人员使用流式细胞仪(FACS)从大量藻类悬浮液中分选出了P1和P2亚群,分选效率超过95%。观察P1和P2亚群的细胞形态发现,P2的生物体积比P1高出1.41倍。这种在nPS暴露下出现的亚群分化现象可能归因于藻类的异质性。长期暴露于NPs的藻类与NPs之间的相互作用可能会触发可遗传的表型多样性和生理特征的分化。在长期NPs暴露后,观察到藻类初级生产力在群体水平上有所提高,而在个体水平上则表现出明显的性状分化,这表明藻类基因组在适应NPs的过程中发生了改变。
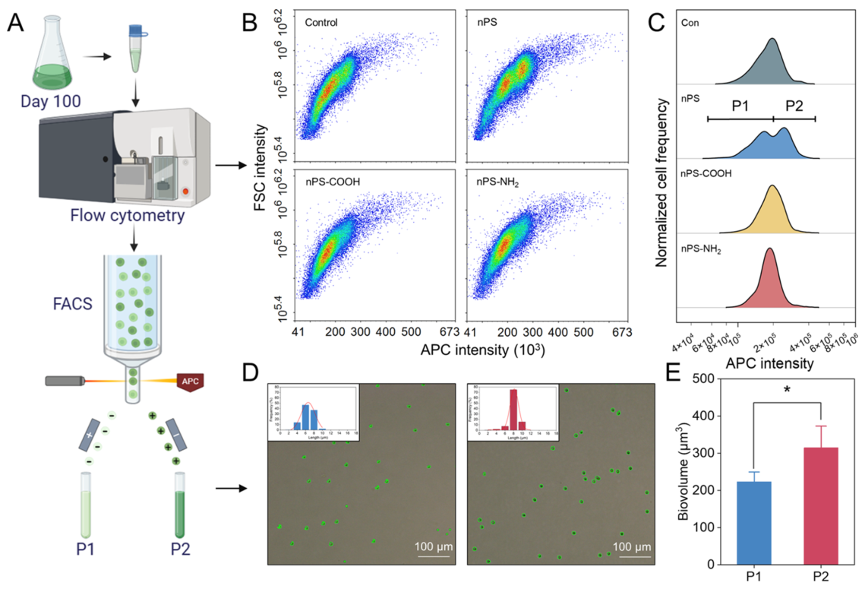
图2:藻类在长期适应纳米塑料(NPs)后的个体水平性状分化。
(A)藻类个体初级生产力检测的示意图。
(B-C)有无NPs处理的藻类细胞在个体水平上的分布情况。
(D)P1和P2的代表性图像及生物体积分布统计。
(E)P1和P2中藻类细胞的生物体积。
(3)NPs长期适应后藻类的全基因组DNA甲基化变化
为全面理解藻类初级生产力的提升,研究人员利用全基因组重亚硫酸盐测序(WGBS)技术,在单碱基分辨率下分析了小球藻在长期暴露于三种NPs后的DNA甲基化图谱,并观察到基因组水平上的变化。在对照组和暴露组中,藻类的甲基化位点数量在10.62M-19.17M,亚硫酸盐转化率高于99%,表明原始数据的完整性和可靠性。从平均甲基化率结果来看,与对照组相比,暴露于nPS、nPS-COOH和nPS-NH₂的藻类发生高甲基化。5-甲基胞嘧啶(5mC)的甲基化水平也呈现出类似的基因组特征。整体甲基化率从对照组的0.88%显著上升至nPS、nPS-COOH和nPS-NH₂暴露组的0.90%、0.89%和0.90%。更多的高甲基化差异甲基化区域(DMRs)导致甲基化水平增加。5mC主要分布在CG位点(对照组为0.83%,nPS为0.84%,nPS-COOH为0.83%,nPS-NH₂为0.84%),其次是CHG和CHH位点。与先前的一项研究类似,无论NPs类型如何,CG位点的甲基化水平和覆盖率均高于CHG和CHH。因此,与CHG和CHH相比,CG位点更适合用于分析在长期NPs暴露下基因组信息的适应性变化。鉴于相邻的甲基化位点通常协同作用,基因表达的变化高度依赖于低甲基化和高甲基化DMRs的水平。
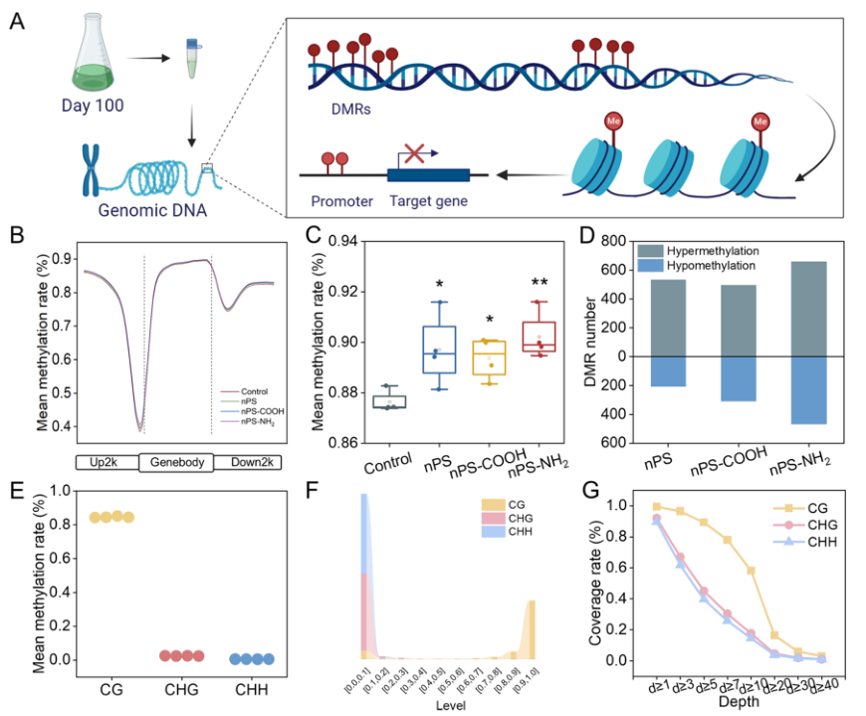
图3:藻类在长期适应纳米塑料(NPs)后的DNA甲基化变化。
(A)DNA甲基化和基因表达的示意图。
(B)5mC在功能基因组区域的相对密度分布。
(C)藻类在有无NPs处理下的平均甲基化率。
(D)暴露于三种NPs后的差异甲基化区域(DMRs)的数量。
(E-G)CG、CHG和CHH的(E)平均甲基化率,(F)甲基化水平,(G)在对照组中的甲基化深度。
(4)藻类DMRs对NPs的适应性变化
研究人员进一步通过比较nPS、nPS-COOH和nPS-NH₂组与对照组的CG位点检测了DMRs。结果显示, nPS暴露组有534个高甲基化DMRs和209个低甲基化DMRs,nPS-COOH暴露组有497个高甲基化DMRs和310个低甲基化DMRs,nPS-NH₂暴露组有660个高甲基化DMRs和470个低甲基化DMRs。因此,在暴露于三种类型的NPs后,高甲基化DMRs的数量均多于低甲基化DMRs。研究人员利用高甲基化区域为每个基因组scaffold构建了甲基化图谱。DMRs在基因组中的分布显示出在每个暴露组的所有支架中均呈现出高甲基化趋势。在DMRs的基因组位点(启动子、外显子、内含子、3'UTR和5'UTR)中,位于启动子和外显子区域的DMRs数量多于内含子区域。任何NPs处理均能诱导更多的高甲基化DMRs位于启动子区域,而在内含子区域则较少。为了阐释DMRs的功能,研究人员利用启动子相关DMRs关联基因(DMGs)进行了KEGG功能富集分析。分析结果表明,暴露于NPs后的高甲基化DMRs和低甲基化DMRs均富集在一系列生物代谢过程(如“光合作用”和“甘油脂代谢”)中,这可能与藻类在长期NPs暴露后初级生产力的提升有关。
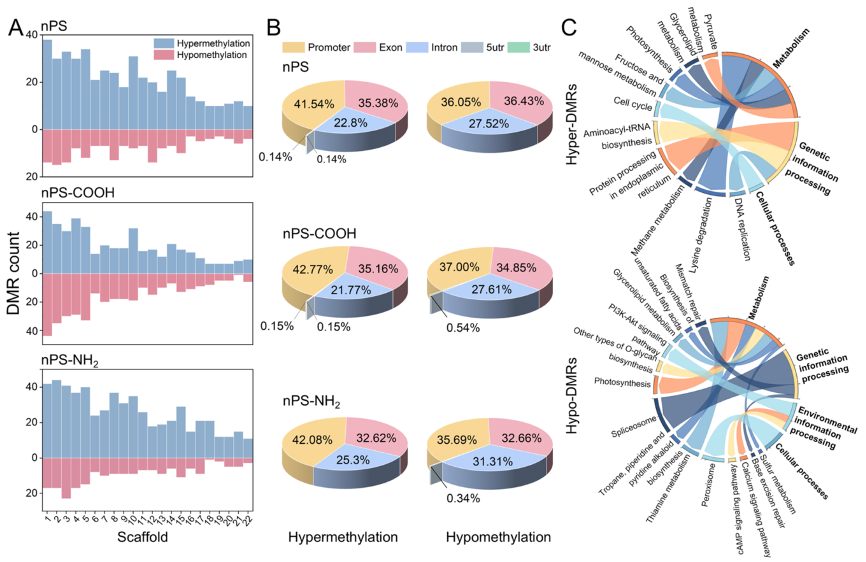
图4:差异甲基化区域(DMRs)的分布与功能。
(A)暴露于三种纳米塑料(NPs)后,DMRs在基因组scaffold中的分布情况。
(B)DMRs在基因组位点(启动子、外显子、内含子、3'UTR和5'UTR)中的分布情况。
(C)在启动子和genebody区域鉴定的DMRs的KEGG功能富集分析。
(5)藻类长期适应后的转录组变化
为将藻类初级生产力的全基因组变化与生理变化联系起来,研究人员利用RNA-seq技术确定了藻类在长期暴露于三种NPs后的转录组变化。转录组结果显示,nPS-COOH暴露组引起的差异表达基因(DEGs)数量显著多于nPS和nPS-NH₂暴露组。此外研究人员还对DEGs进行注释,并进行基因集富集分析(GSEA)以研究转录组变化功能。一系列与氨基酸和肽链合成相关通路(如tRNA生物合成、核糖体和核糖体生物合成)被三种类型的NPs抑制,表明所有三种类型NPs都能干扰蛋白质合成。外泌体通路被所有三种NPs上调,这一过程有助于从衰老细胞中清除ROS、氧化脂质和其他有害代谢物,从而重新启动藻类增殖。光合作用蛋白通路在nPS-NH₂处理下显著下调。这一结果与DMRs富集结果一致(图4C),可能解释了为什么在nPS-NH₂处理后藻类生物量和生物体积没有显著变化(图1E、F)。
(6)NPs长期适应后基因组变化与基因表达水平的关系
研究人员进一步分析了甲基化组和转录组的结果,以揭示藻类在长期NPs暴露下受到影响的分子机制。DMGs和DEGs的整合分析结果揭示了nPS-COOH的DEGs数量多于其他两组,因此在nPS-COOH暴露组中鉴定出的候选基因最多(69个)。本研究通过启动子相关DMRs关联基因(PDMGs)与DEGs筛选出的候选基因富集于一系列通路,如蛋白质代谢过程、蛋白水解和含氮有机化合物代谢过程。相比之下,通过genebody(基因体)相关DMRs关联基因(GDMGs)与DEGs筛选出的候选基因富集于催化活性的正向调控、分子功能和水解酶活性等通路。这一结果表明碳和氮的代谢受启动子区域甲基化调控。这些通路的调控可能代表了藻类对长期NPs暴露的适应性响应,并进一步影响下游功能。基因表达与DNA甲基化之间的Pearson相关性分析结果表明,PDMGs在nPS和nPS-NH₂暴露组中与DEGs呈负相关,在nPS-COOH暴露组中则呈正相关。而GDMGs在nPS和nPS-NH₂暴露组中与DEGs呈正相关,但在nPS-COOH暴露下则呈负相关。先前研究已经表明,启动子区域高甲基化和基因体区域的低甲基化会抑制目标基因表达,而启动子区域低甲基化和基因体区域高甲基化则会促进目标基因表达。nPS和nPS-NH₂暴露组中DMRs和DEGs的相关性与先前的研究结果一致,但nPS-COOH暴露组的相关性则与先前研究结果相反。DNA甲基化对转录调控的负调控方向表明,nPS-COOH可能会导致藻类基因转录沉默,而nPS和nPS-NH₂则不会。这表明在长期暴露于不同电荷的NPs后,藻类在基因组水平上的适应策略存在差异。
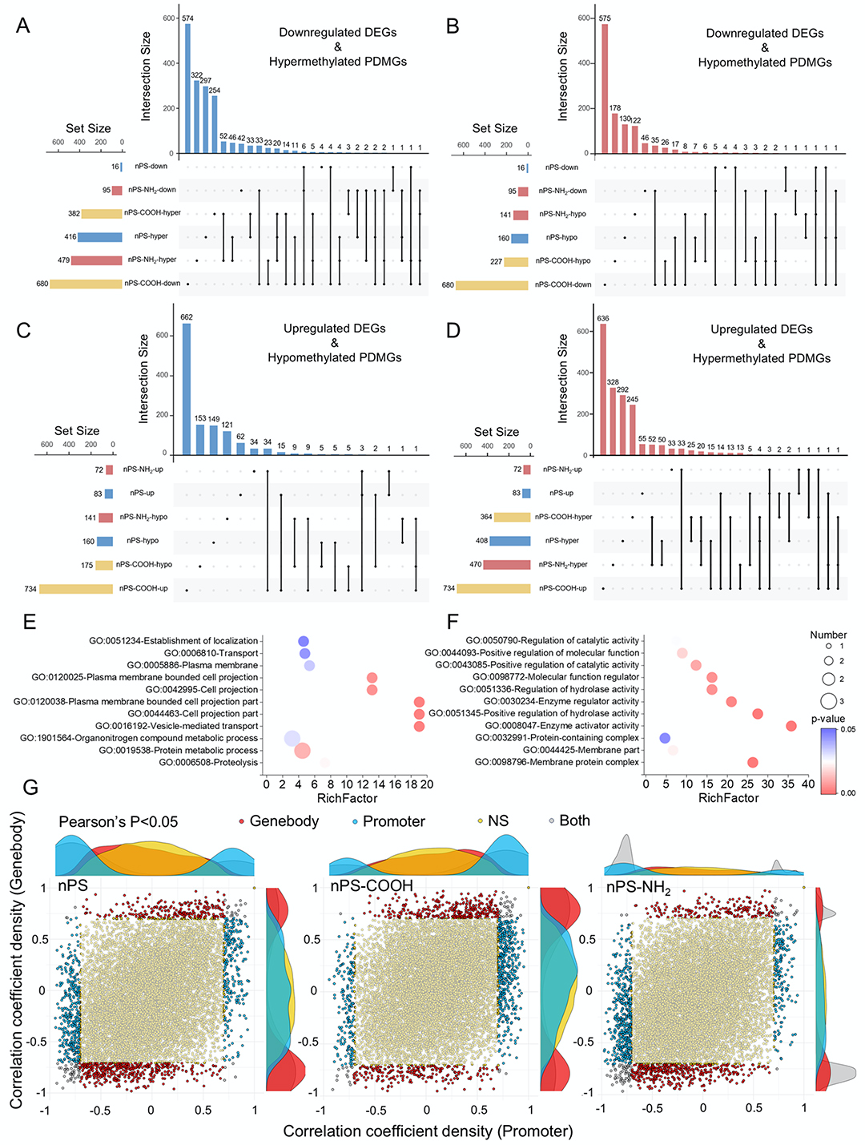
图5:长期NP适应后基因组变化与基因表达水平的整合分析结果。
(A) 下调的差异表达基因(DEGs)与启动子相关高甲基化差异甲基化区域(DMRs)关联基因(PDMGs)。
(B) 下调的DEGs与基因体相关低甲基化DMRs关联基因(GDMGs)。
(C) 上调的DEGs与低甲基化PDMRs。
(D) 上调的DEGs与高甲基化GDMRs。
(E) 对于(A)和(C)中的候选基因进行GO功能富集分析;
(F) 对于(B)和(D)中的候选基因进行GO功能富集分析;
(G) 暴露于三种NPs后,每个基因的基因表达与DNA甲基化之间的Pearson相关性分析。
参考文献:
Liu S, Huang X, Han J, Yao L, Li H, Xin G, Ho SH, Zhao J, Xing B. Genome-Wide Molecular Adaptation in Algal Primary Productivity Induced by Prolonged Exposure to Environmentally Realistic Concentration of Nanoplastics. ACS Nano. 2024 Oct 19. doi: 10.1021/acsnano.4c09709.