细菌微生物-染色质免疫共沉淀测序(ChIP-seq)
染色质免疫共沉淀(Chromatin Immunoprecipitation,ChIP),是研究体内蛋白质与DNA相互作用的经典方法。将ChIP与高通量测序技术相结合的ChIP-Seq技术,可在全基因组范围对特定蛋白的DNA结合位点进行高效而准确的筛选与鉴定,为研究的深入开展打下基础。
DNA与蛋白质的相互作用与基因的转录、染色质的空间构型和构象密切相关。运用组蛋白特定修饰的特异性抗体或DNA结合蛋白或转录因子特异性抗体富集与其结合的DNA片段,并进行纯化和文库构建,然后进行高通量测序,通过将获得的数据与参考基因组精确比对,研究人员可获得全基因组范围内某种修饰类型的特定组蛋白或转录因子与基因组DNA序列之间的关系,也可对多个样品进行差异比较。
DNA与蛋白质互作(ChIP):提供组蛋白修饰、转录因子结合等抗体富集、建库测序、数据分析全流程服务。具有细胞,动、植物组织,微生物(弓形虫、烟粉虱等)等不同样本富集建库经验。
技术优势:
1)物种范围广:细胞、动物组织、植物组织、细菌微生物多物种富集经验;
2)微量建库:只需5ng以上免疫沉淀后的DNA,即可展开测序分析;
3)方案灵活:根据不同的项目需求,选择不同的组蛋白修饰特异性抗体。
实验策略:
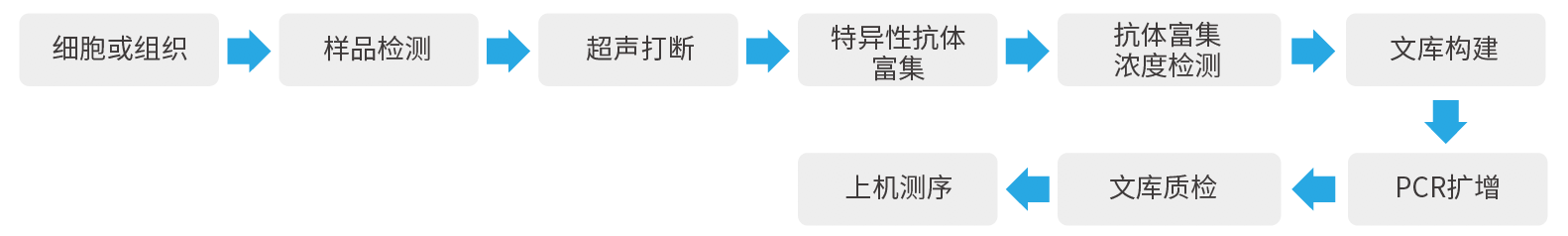
分析内容:
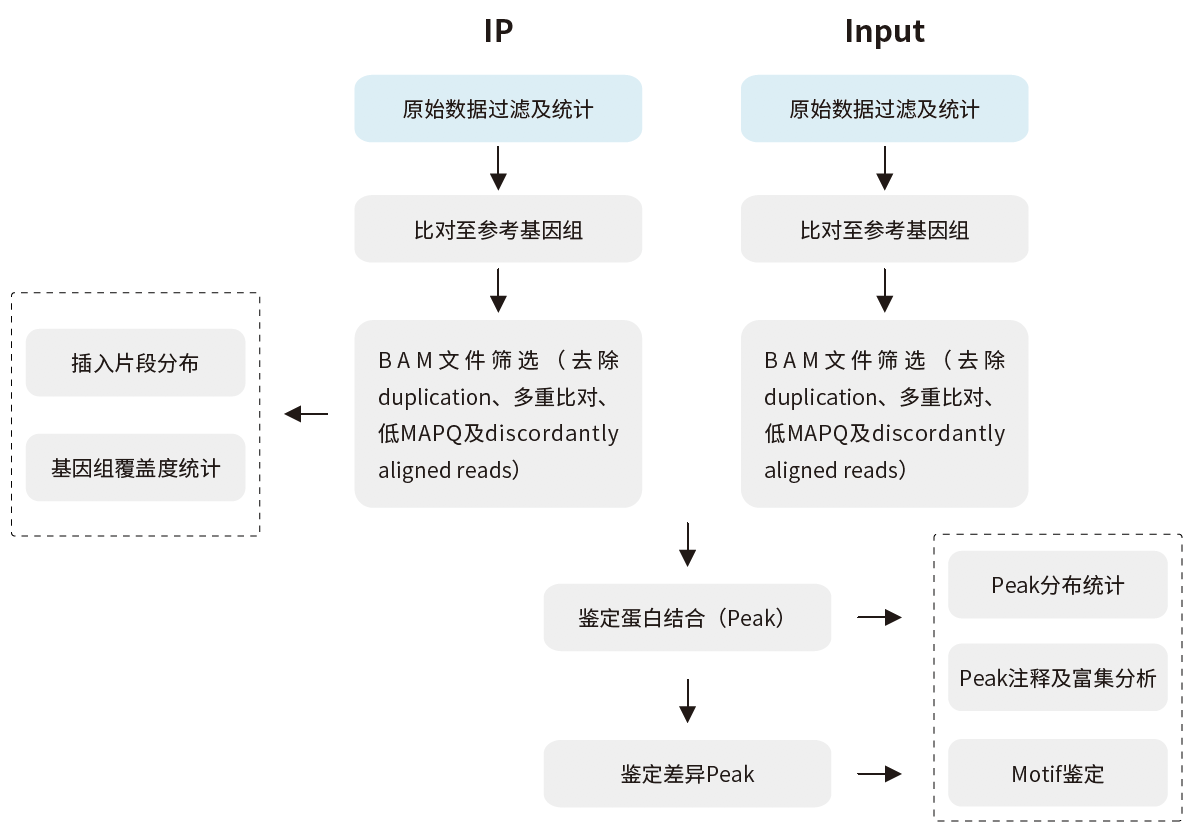
送样要求:

项目案例:
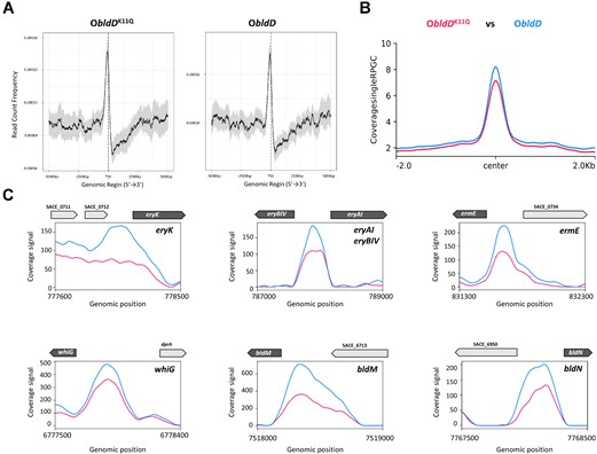
标题:A meet-up of acetyl phosphate and c-di-GMP modulates BldD activity for development and antibiotic production
发表杂志:Nucleic Acids Res
影响因子:14.6
发表年份:2023
(2)微生物:烟粉虱
标题:A bacteriocyte symbiont determines whitefly sex ratio by regulating mitochondrial function
发表杂志:Cell Reports
影响因子:8.8
发表年份:2023
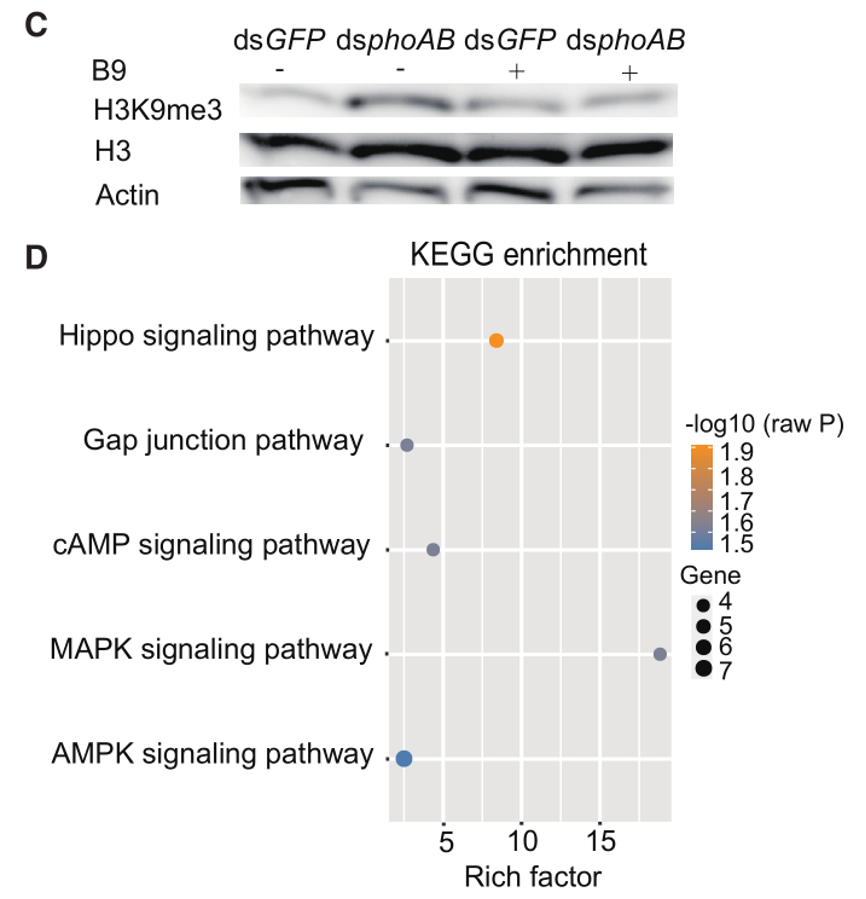
使用H3K9me3抗体对±HBt烟粉虱进行ChIP-seq分析
(3)细胞:小鼠
标题:ARTICLE JMJD3 acts in tandem with KLF4 to facilitate reprogramming to pluripotency
发表杂志:Nat Commun
影响因子:16.6
发表年份:2020
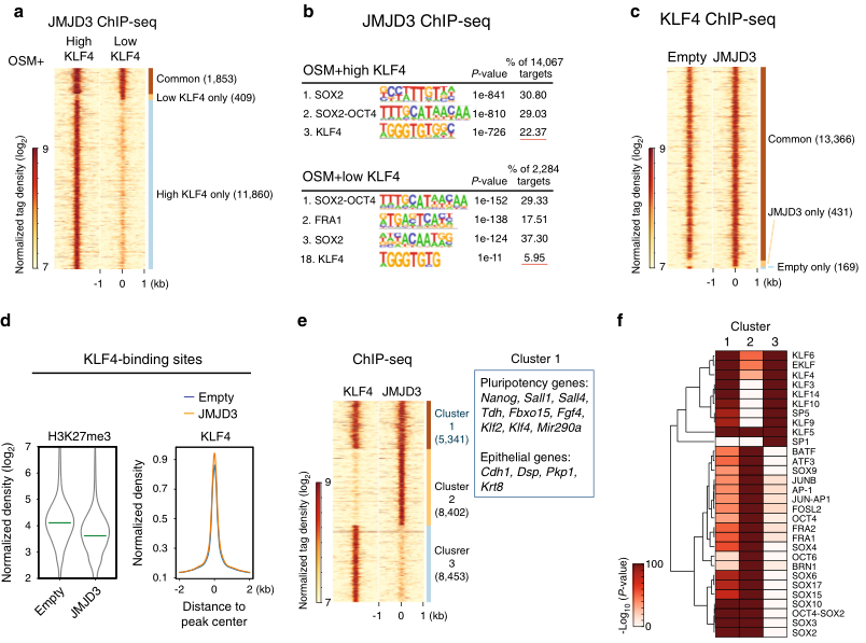
ChIP-seq揭示小鼠细胞重编程过程中JMJD3与KLF4协同作用
参考文献:
① Fu Y,et al. A meet-up of acetyl phosphate and c-di-GMP modulates BldD activity for development and antibiotic production. Nucleic Acids Res. 2023 Jul 21;51(13):6870-6882.
② Yao YL,et al. A bacteriocyte symbiont determines whitefly sex ratio by regulating mitochondrial function. Cell Rep. 2023 Feb 10;42(2):112102.
③ Huang Y,et al.JMJD3 acts in tandem with KLF4 to facilitate reprogramming to pluripotency. Nat Commun. 2020 Oct 8;11(1):5061.
