羟甲基化-全基因组精准甲基化/羟甲基化测序(oxWGBS+WGBS)
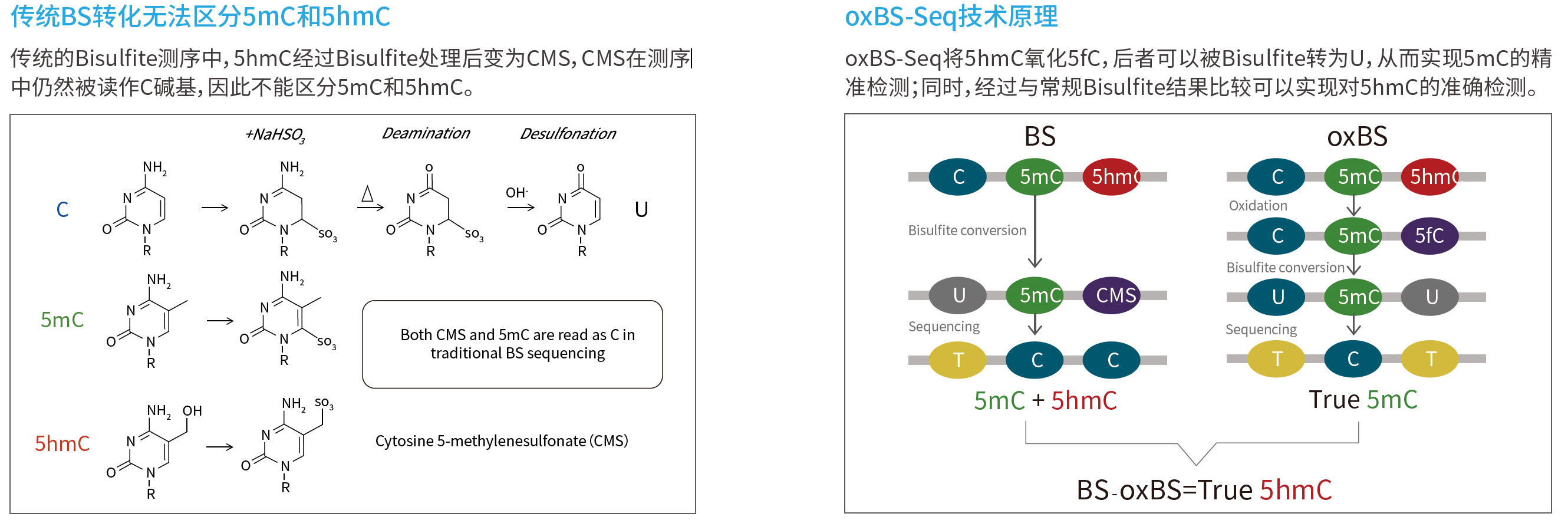
羟甲基化5hmC是哺乳动物基因组上的第六碱基,在发育、衰老、神经退行性疾病、复杂疾病及肿瘤发生过程中起重要作用。DNA羟甲基化是近年发现的一种新的DNA修饰并迅速成为研究热点。随着研究的深入,发现之前被认为是检测DNA甲基化标准的重亚硫酸盐测序并不能区分DNA甲基化(5mC)和DNA羟甲基化(5hmC)。
易基因联合剑桥大学建立了化学氧化法结合重亚硫酸盐转化的测序技术(oxidative bisulfite sequencing, oxBS-Seq),该技术不仅可以精确检测DNA甲基化,排除DNA羟甲基化的影响,还可以双文库结合同时单碱基分辨率精确检测DNA羟甲基化。
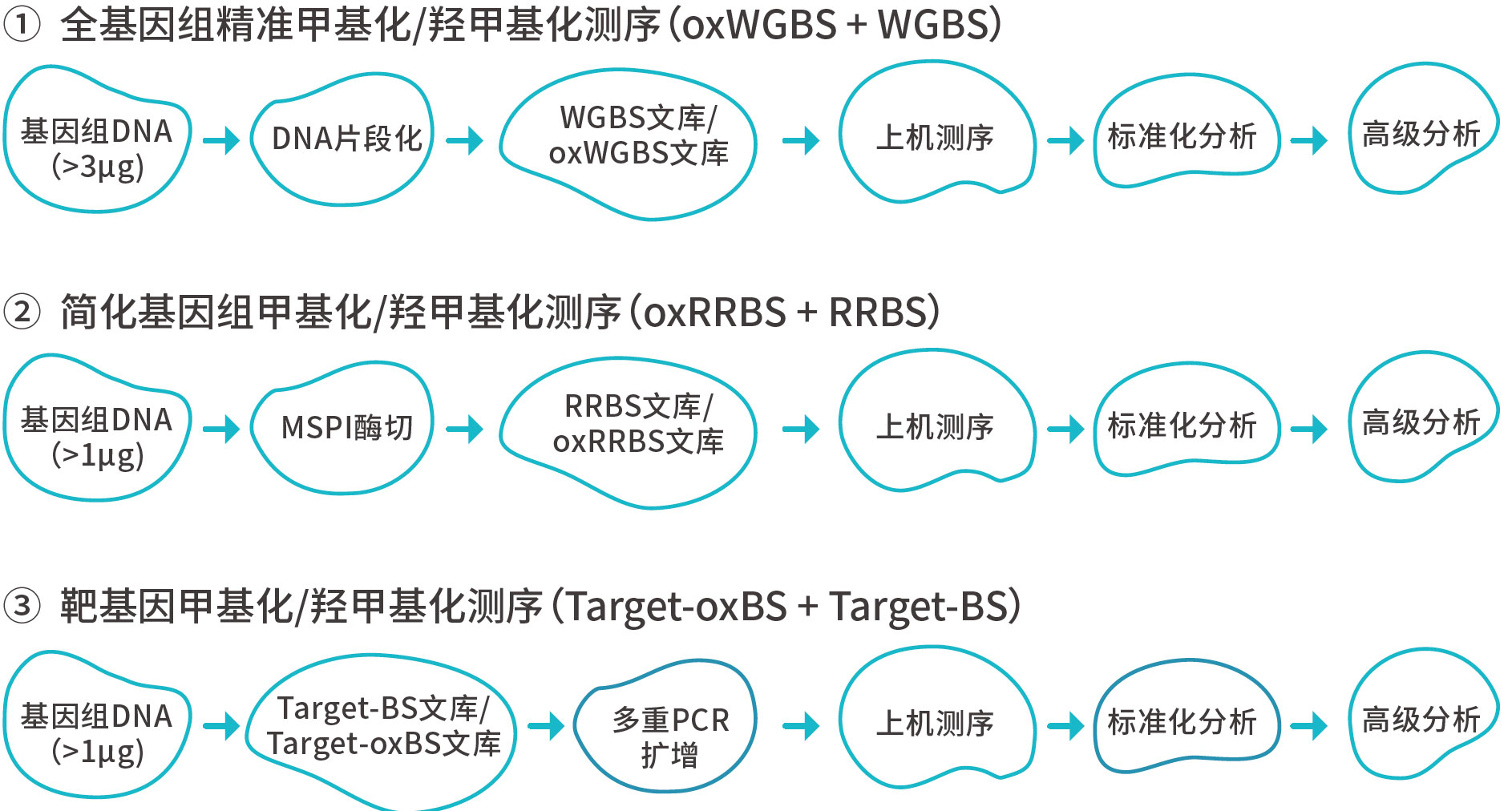
技术路线
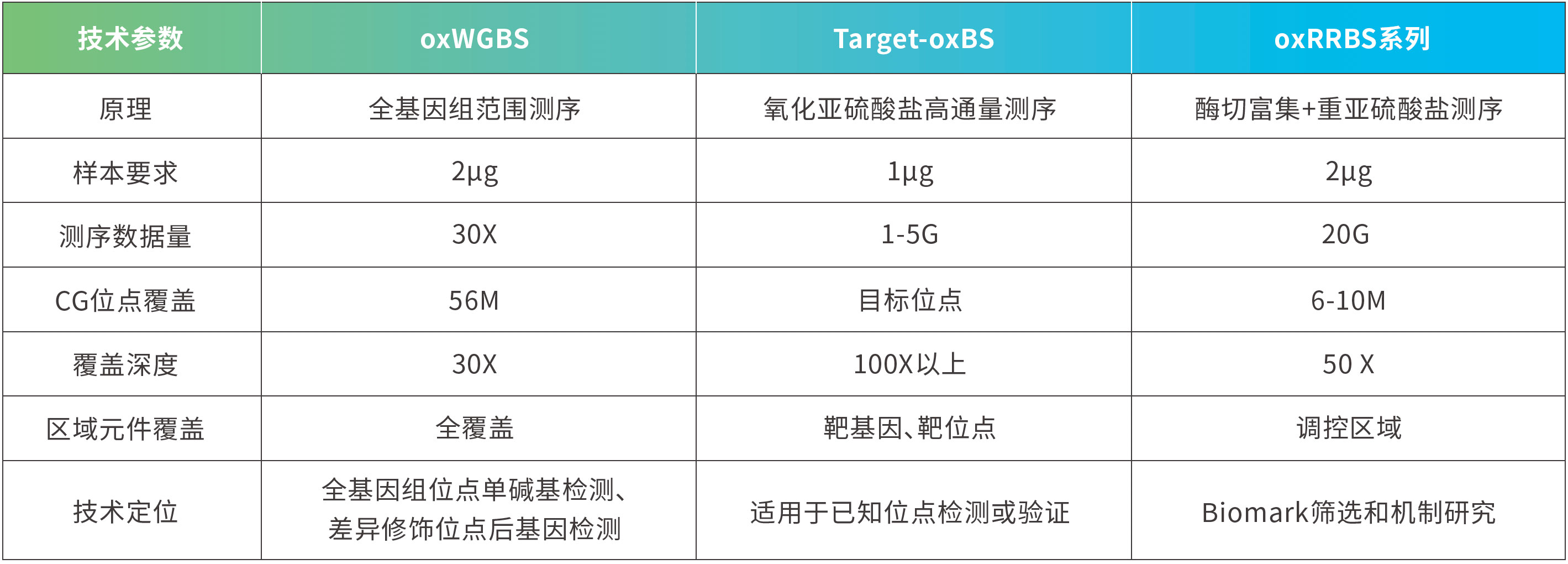
技术指标
技术优势:
1、DNA甲基化检测全新的“标准”;
2、单碱基检测DNA羟甲基化修饰;
3、多重质控标准检测氧化效率和Bisulfite转换率;
4、实验偏好性低,重复性高(R2>0.98);
5、易基因自主研发的甲基化特异性多重PCR引物设计软件;
6、可满足多种测序应用需求:
(1)全基因组氧化甲基化测序(oxWGBS)
(2)简化基因组氧化甲基化测序(oxRRBS)
(3)目标区域靶基因氧化甲基化测序(Target-oxBS)。
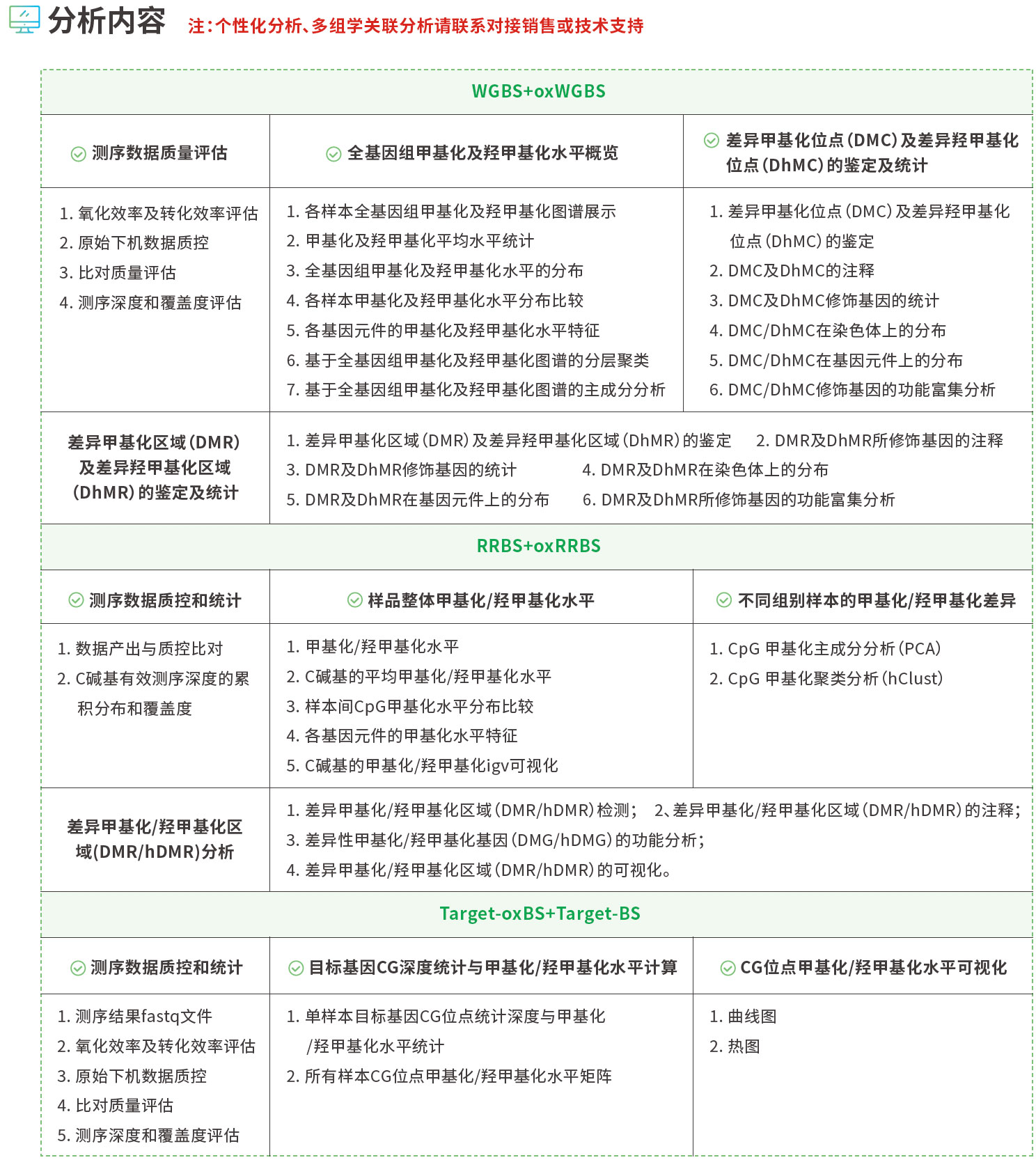
分析内容
样本及送样要求
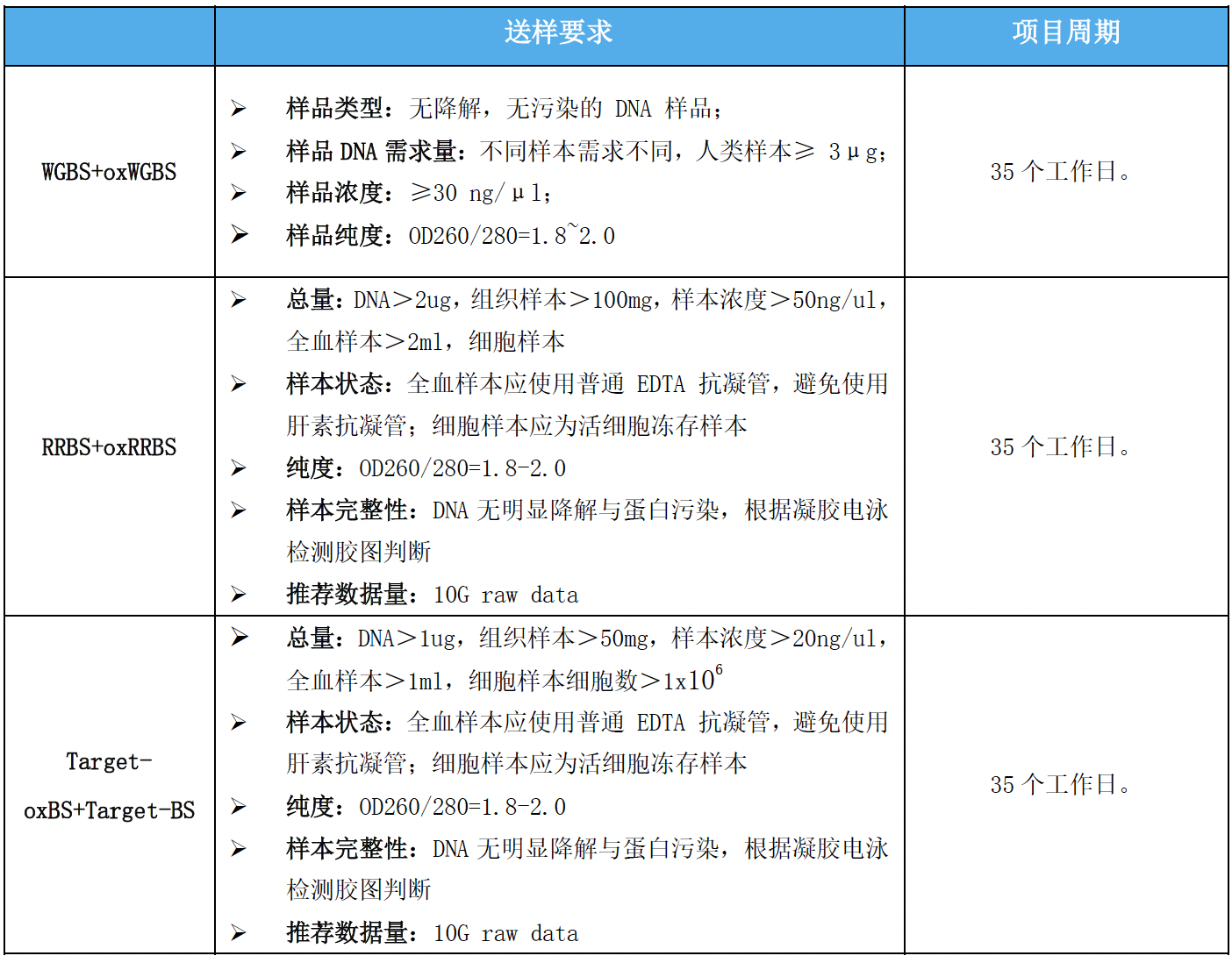
经典案例
WGBS+oxWGBS项目文章:
An epigenomic landscape of cervical intraepithelial neoplasia and cervical cancer using single-base resolution methylome and
hydroxymethylome. (单碱基oxBS-seq宫颈上皮内瘤变和宫颈癌进行表观遗传学研究)
1、背景:
有许多研究表明宫颈癌的发生和进展跟异常的表观遗传修饰相关,主要包括胞嘧啶甲基化(5mC)和羟甲基化(5hmC),通过5mC/5hmC对基因表达的表观遗传调控在肿瘤发生过程中起着关键作用。然而这些研究没有绘制完整的表观基因图谱,不能从健康宫颈到宫颈上皮内瘤变(CIN)再到宫颈癌(CC)整个发病机制中区分5mC和5hmC。另外通过多组学综合分析在宫颈癌中鉴定生物标志物的报道数量十分有限。
2、方法:
本文作者选择从健康宫颈到CIN到CC的一系列样本,进行全基因组亚硫酸氢盐测序 (WGBS-seq)、oxWGBS-seq、RNA-seq 和组蛋白修饰数据进行综合分析,以确定CC特异性的潜在表观遗传生物标记物。
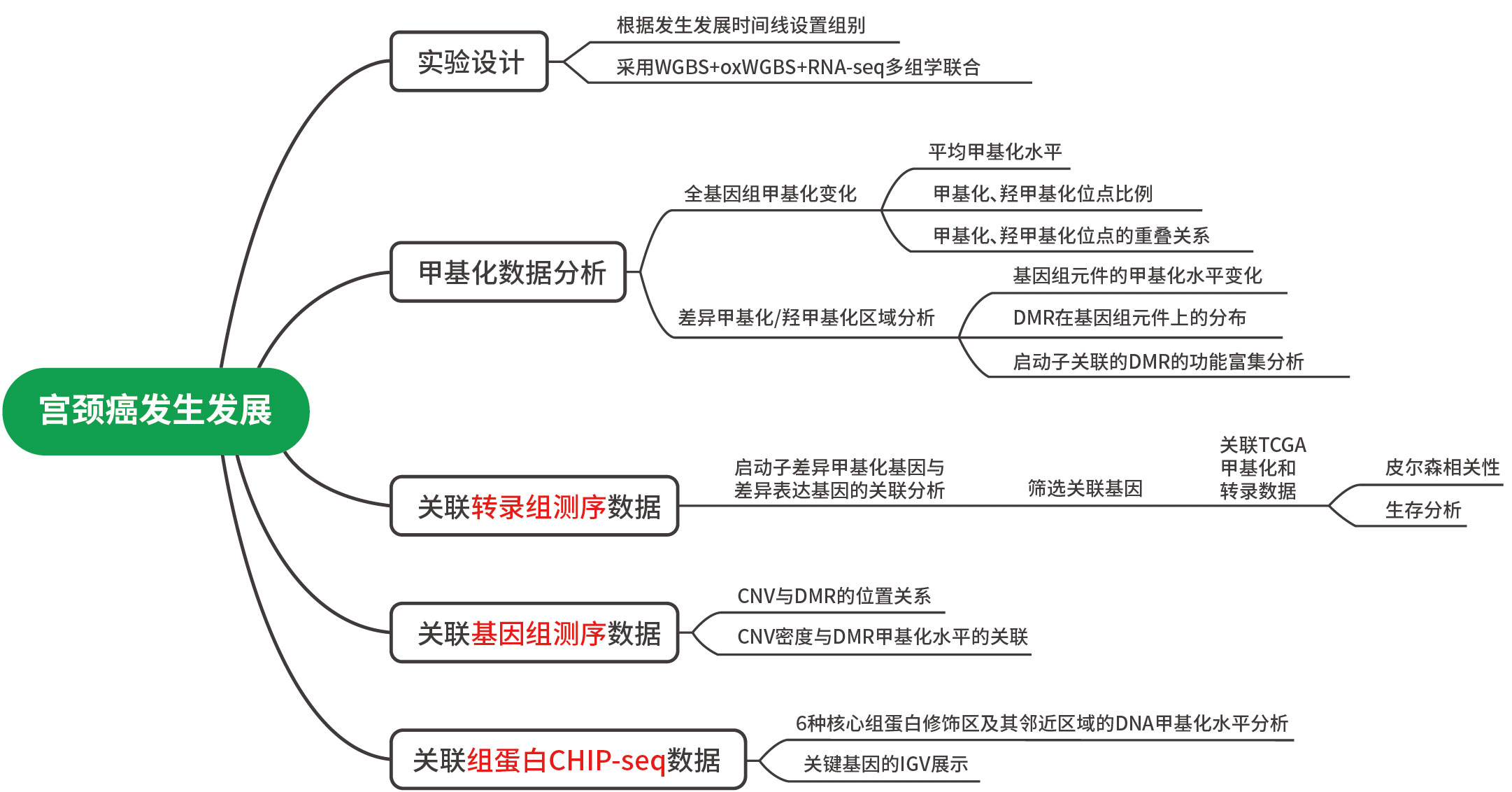

3、结论:
本研究采用oxBS-seq方法绘制从健康宫颈到CIN到CC样本在单碱基分辨率下的全基因组5mC/5hmC图谱。分析CC进展过程中DMR和DhMR区域变化趋势。GO/KEGG富集分析与DMRs相关基因 (DAGs) 和DhMRs相关基因 (DhAGs)。个性化分析DAGs和DhAGs甲基化和羟甲基化水平与基因表达的关系。多组学综合分析甲基化/羟甲基化与基因组变异的相关性。对甲基化/羟甲基化与组蛋白修饰关联分析。通过多组学分析证实了甲基化/羟甲基化与宫颈癌发生中的 CNV 无关,且甲基化/羟甲基化的发生远早于CNV。确定了八个预后相关基因,可以作为CC治疗的新靶点,这为表观遗传学在肿瘤发生和进展中的筛查诊断及预后提供了重要数据支持。
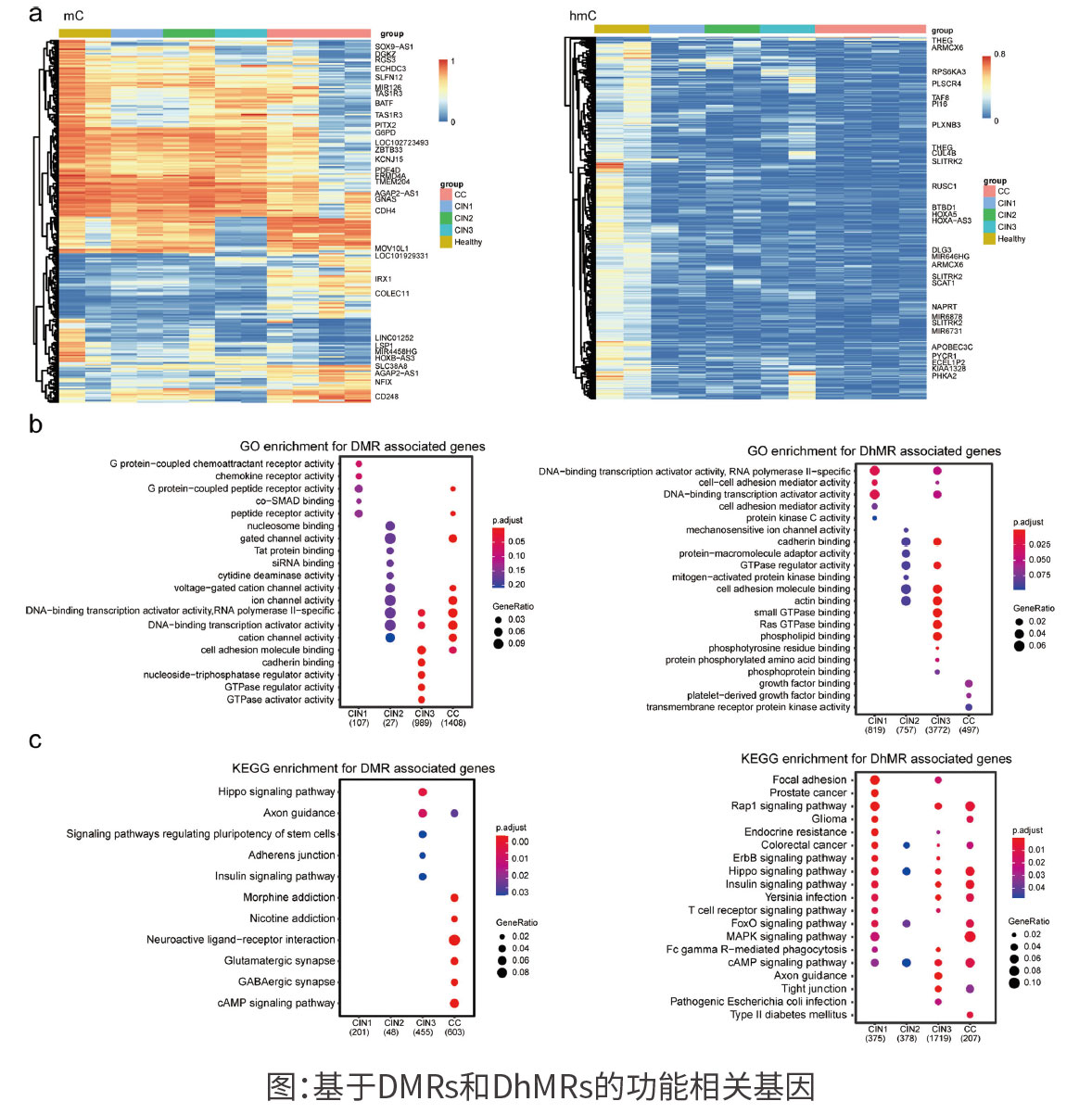
RRBS+oxRRBS医学方向案例:
oxRRBS+RRBS单碱基水平检测炎症性肠炎(IBD)小鼠模型的结肠组织DNA甲基化和羟甲基化
Han Q,et al.Multi-Omics Characterization of Inflammatory Bowel Disease-Induced Hyperplasia/Dysplasia in the Rag2-/-/Il10-/-Mouse Model. Int J Mol Sci. 2020 Dec 31;22(1) pii: ijms22010364.
1、背景:
多项研究表明炎症性肠炎(IBD)中存在基因组DNA甲基化失调,然而对DNA羟甲基化水平变化则关注有限。此前的研究未能分别绘制5hmC和5mC这两种表观遗传标记,限制了对IBD和结直肠癌(CRC)表观遗传机制的理解。
2、方法:
研究利用RRBS+oxRRBS分别绘制了Rag2-/-/Il10-/-H.hepaticus感染的IBD小鼠模型近端结肠组织DNA甲基化和羟甲基化图谱,进行DNA甲基化/羟甲基化差异分析和相应的转录组关联分析。
3、结论:
本研究利用RRBS+oxRRBS技术鉴定出1606个差异甲基化区域(DMR)和3011个差异羟甲基化区域(DhMR)。这些DMR/DhMR与胃肠道疾病、炎症性疾病和癌症相关的基因重叠。研究表明慢性炎症会引发基因组甲基化和羟甲基化模式改变,肿瘤发生的关键基因表达变化,并可能导致结直肠癌发生。
RRBS+oxRRBS农学项目文章:
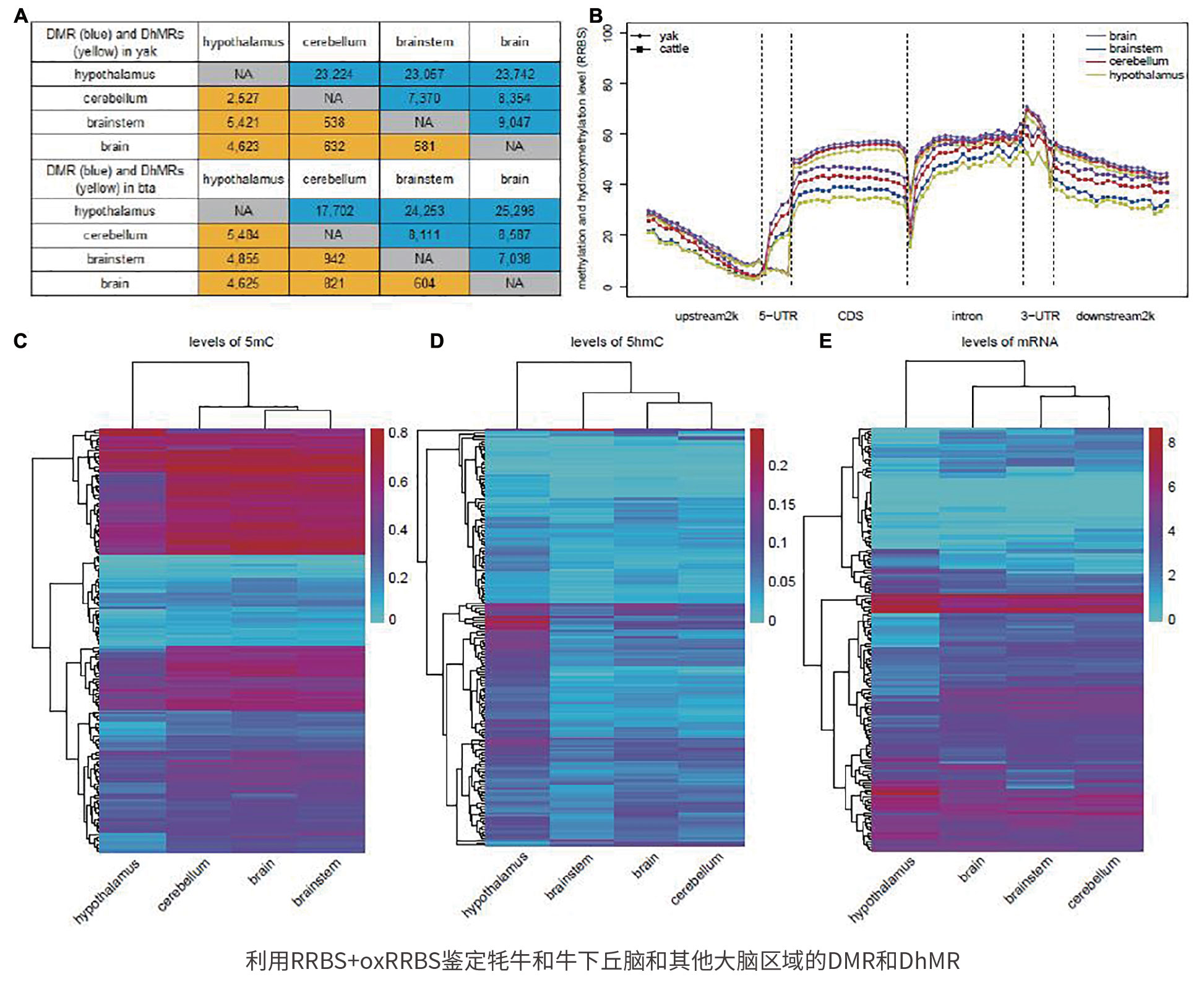
oxRRBS+RRBS揭示了牦牛下丘脑在神经调节和髓鞘形成中的表观调控作用
Chai Z, et al. Genome-Wide DNA Methylation and Hydroxymethylation Changes Revealed Epigenetic Regulation of Neuromodulation and Myelination in Yak Hypothalamus. Front Genet. 2021;12:592135.
1、背景:
5-甲基胞嘧啶 (5mC) 和 5-羟甲基胞嘧啶 (5hmC) 都是神经发育中重要的表观遗传修饰。然而很少有研究在自然高海拔条件下鉴定动物大脑区域的全基因组5mC和5hmC模式。
2、方法:
利用RRBS+oxRRBS鉴定牦牛和牛的大脑、脑干、小脑、下丘脑的基因组5mC和5hmC位点,绘制基因组DNA甲基化和羟甲基化图谱,并进行DNA甲基化/羟甲基化差异化分析和对应转录组的关联分析。
3、结论:
本研究利用RRBS+oxRRBS技术发现牦牛和牛的下丘脑和其他大脑区域的5mC和5hmC存在显著差异,鉴定出差异甲基化区域(DMR)和差异羟甲基化区域(DhMR),其中大多数彼此重叠。最后,验证了DMRs和DhMRs调控的差异表达基因(DEG)可能在神经调节和髓鞘形成中发挥重要作用。总之结果表明, 5mC和5hmC介导的表观遗传调控可能广泛影响下丘脑的发育及其生物学功能,可能有助于提高高海拔条件的生理适应性。
参考文献:
①Han Y,et al.An epigenomic landscape of cervical intraepithelial neoplasia and cervical cancer using single-base resolution methylome and hydroxy methylome. Clin Transl Med. 2021 Jul;11(7):e498.
②Han Q,et al.Multi-Omics Characterization of Inflammatory Bowel Disease-Induced Hyperplasia/Dysplasia in the Rag2-/-/Il10-/- Mouse Model. Int J Mol Sci. 2020 Dec 31;22(1) pii: ijms22010364.
③Chai Z, et al. Genome-Wide DNA Methylation and Hydroxymethylation Changes Revealed Epigenetic Regulation of Neuromodulation and Myelination in Yak Hypothalamus. Front Genet. 2021;12:592135.
