微生物-扩增子测序(16S/18S rDNA,ITS)
扩增子测序主要是针对有一定物种区分度的基因片段的PCR产物进行测序分析。针对不同的研究目的和需求,易基因提供三种扩增子测序产品:16S rDNA测序、18S rDNA测序、ITS测序。
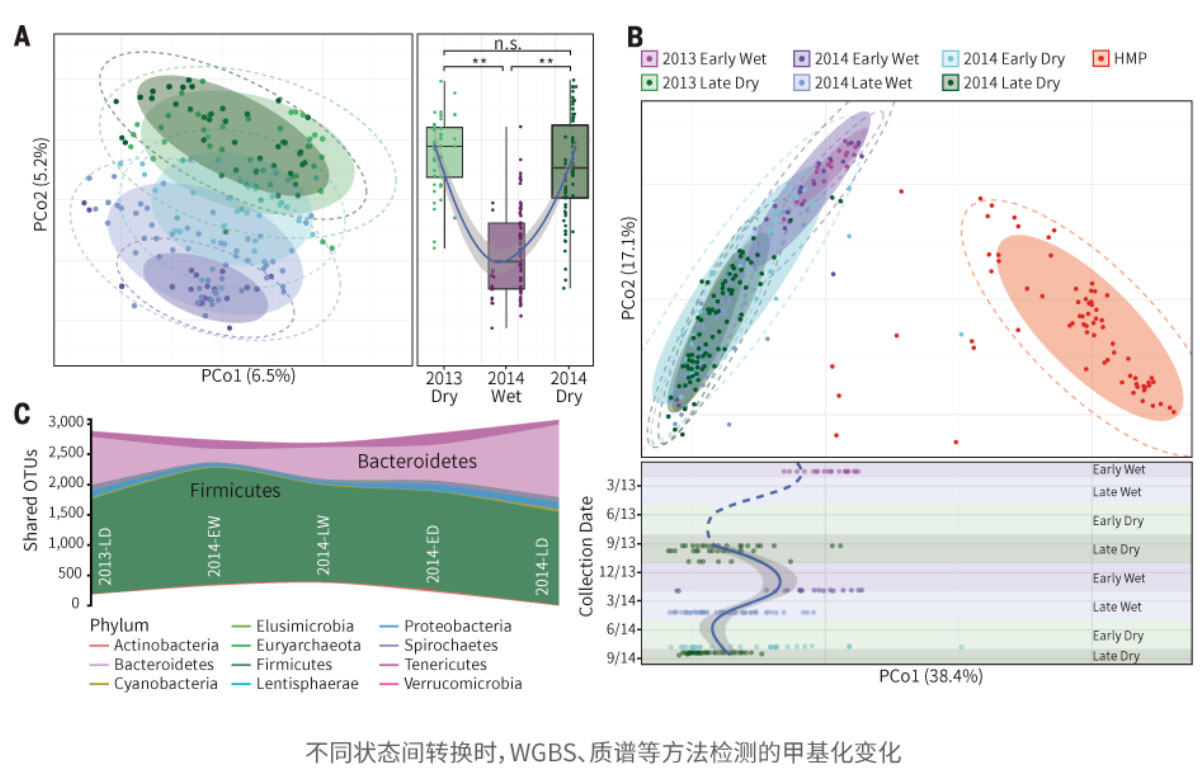
16S rDNA测序:16S rDNA为原核生物核糖体小亚基rRNA的DNA编码序列,结构稳定,具有9个高可变区(下图, v1-v9)和10个保守区。高可变区具有良好的物种特异性,对若干高可变区进行测序,可以在一定分类水平上解析细菌或者古菌的群落结构多样性。
技术优势
实验策略
信息分析

送样要求

研究案例
坦桑尼亚哈扎人狩猎采集者肠道微生物的季节变化
Smits S A, Leach J, Sonnenburg E D, et al. Seasonal cycling in the gut microbiome of the Hadza hunter-gatherers of Tanzania.[J]. Science, 2017, 357(6353):802-806
背景:
坦桑尼亚的哈扎人是世界上仅存的狩猎部落,以原始的狩猎和采集方式生存繁衍。哈扎人的饮食结构比较单一,未受到现代工业文明的影响。他们的饮食结构在旱季主要以狩猎和植物块茎为食,在雨季会更多的食用蜂蜜和浆果,季节性特征相当显著。这种饮食结构导致他们的肠道微生物菌群易于对食物中的粗纤维进行消化。之前的研究也表明,哈扎人男性与女性由于分工不同,体内的肠道微生物菌群也有很大不同。肠道菌群帮助人类在数百万年的进化过程中适应对不同种类食物的消化。本研究以哈扎人狩猎采集者肠道菌群为研究对象,其中一部分工作是利用16S rDNA测序进行菌群结构季节性周期变化规律的分析。
方法:
该研究在不同季节收集了来自188个哈扎人的350个粪便样本及饮食数据利用16SrDNA测序技术进行菌群分析。
结论:
作者对哈扎人肠道微生物菌群进行分析,发现哈扎人肠道微生物菌群具有周期性和季节不同性。其中,在旱季和雨季的间隙,哈扎人肠道中70%的拟杆菌会消失,但在这段时间之后,这些拟杆菌会重新出现。在哈扎人的肠道中,Prevotellaceae,Spirochaetes,Succinivibrionaceae,Lachnospiraceae四个科的细菌受季节改变最为显著。同时,将哈扎人肠道微生物菌群与来自于16个国家18个人群的微生物菌群进行比较,发现工业化地区拟杆菌在菌群中占比21%,显著高于传统食物地区拟杆菌在菌群中占比0.8%。